Difyで作る論⽂仕分けアプリpart2: PubMedAPIから詳細を取得
| 目次 |
1. はじめに
本記事は、Difyのチャットワークフローを使ってPubMed論⽂の検索‧翻訳‧要約を⾃動化するシリーズのPart 2です。
Part 1の振り返り:
- ⾃然⾔語クエリ(⽇本語)からPubMed検索パラメータを抽出
- E-SearchでPMIDリストを取得
- 後段ノードに渡すためPMIDをカンマ区切り⽂字列へ整形
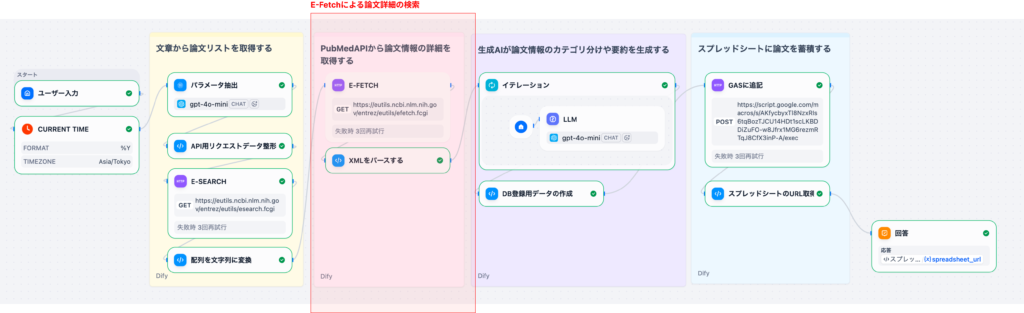
Part 2(本記事)では、PMIDリストをもとにE-Fetchで論⽂の詳細データを取得し、後続のAI処理で扱いやすい構造化データへ変換するまでを解説します。ここでは、PubMedから返却されるXMLのパース処理を丁寧に解説します。
シリーズ構成
- Part0: 全体像とPubMed API基礎
- Part 1: パラメータ抽出とE-Search編
- Part 2(本記事): E-Fetchとデータパース編
- Part 3: AI処理‧データ整形編
- Part4: データ保存とGAS連携編
2. ワークフローの位置づけ
Part 1で整形したPMID⽂字列は、これから紹介するノードに渡されます。
- E-Fetch: 論⽂の詳細データを取得(XML形式)
- XMLパース: LLM処理で扱いやすいPython dict / list形式へ変換
この簡易版では、E-Fetchのみを使⽤して論⽂の詳細データを取得します。E-Summaryは使⽤せず、常にE-FetchでXML形式のデータを取得することで、アブストラクトやMeSH⽤語などの詳細情報を確実に取得できます。
ここまでを整えることで、Part 3で実施するAI要約‧優先度付けをスムーズに実装できます。
3. 各ノードの詳細解説
3-1. E-Fetch(HTTP Requestノード)
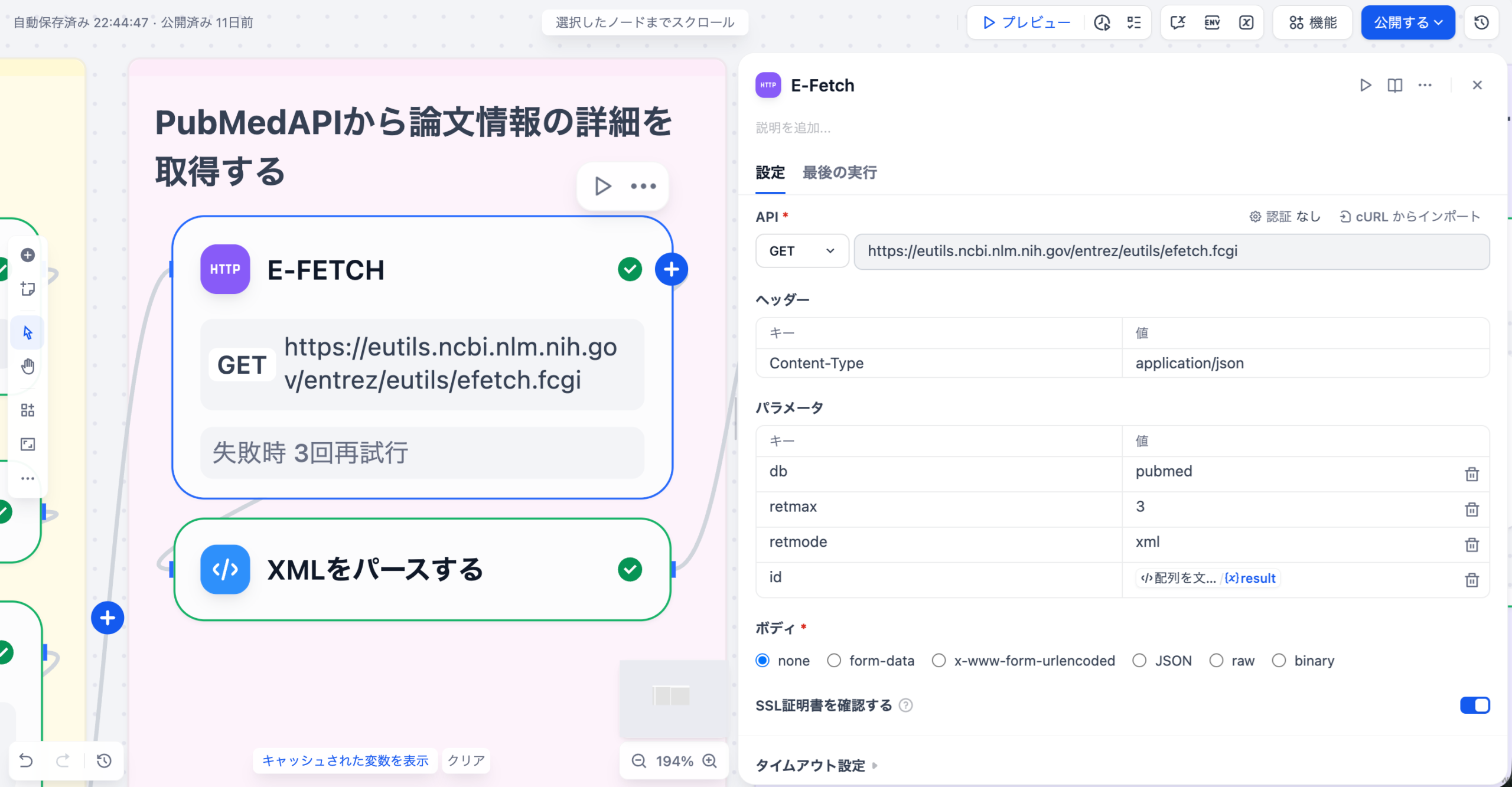
PubMedの E-fetch APIを呼び出して、論⽂の詳細データをXML形式で取得するノードです。
この簡易版のワークフローでは、テスト⽤に retmax を固定で 3 に設定することで、最大取得件数を3件に抑えています。
| パラメータ | 値 | 説明 |
|---|---|---|
| URL | https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi | PubMed E-Fetchエンドポイント |
| メソッド | GET |
パラメータ
| パラメータ | 値 | 説明 |
|---|---|---|
| db | pubmed | データベース名 |
| id | {{#1764077943290.result#}} | カンマ区切りのPMID⽂字列(前のノードから取得) |
| retmode | xml | XMLレスポンスを取得 |
| retmax | 3 | 取得件数(テスト⽤に3件で固定) |
特徴: Abstract、MeSH、著者、掲載誌など詳細なデータがすべて含まれるため、LLM要約やキーワード抽出に最適です。
3-2.XMLレスポンスのパース(Codeノード)
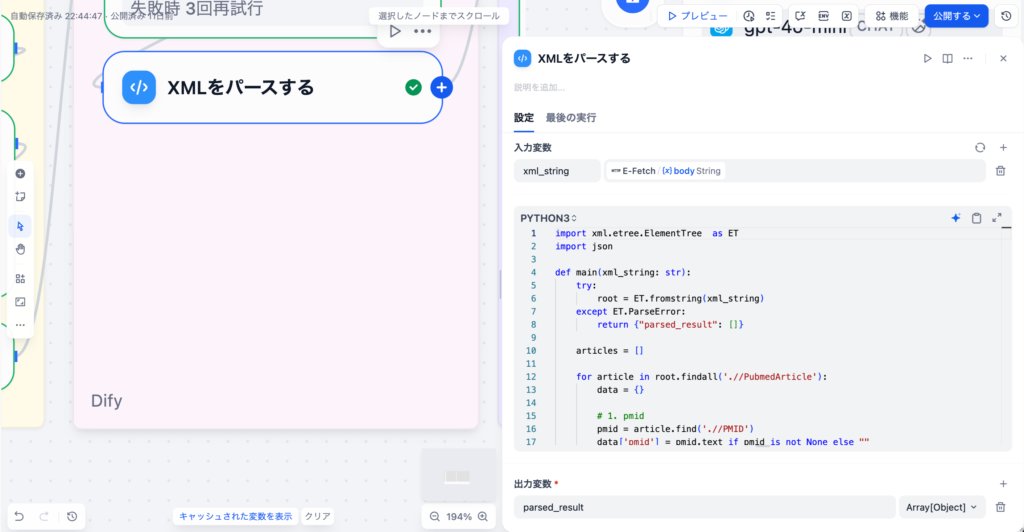
E-FetchのエンドポイントはXML形式のデータを返します。
通常のJSON文字列ではないため、コードブロックにてPythonのxml.etree.ElementTree を使って必要な項⽬を抽出・整形します。
(Difyでは標準的なPythonライブラリを呼び出して使うことができます)
以下のコードは理解していなくても⼤丈夫です。コピペで動かすことができます(コードブロックの⼊⼒変数と出⼒変数の名称や、タイプを揃えるよう注意してください)
主な抽出項⽬
| 項⽬ | 説明 |
|---|---|
| pmid | PubMed ID |
| title | 英語タイトル |
| abstract | AbstractTextに付与されたLabel込みで抽出し、改⾏で結合 |
| author | 著者⼀覧(”Forename Lastname”形式) |
| main_author_affiliation | 第⼀著者の所属機関 |
| journal_inshort | 雑誌略称 |
| journal | 雑誌正式名称 |
| year | 公開年( PubDate → DateCompleted の順に参照) |
| doi | DOI(ELocationIDから抽出) |
| MeSH_Keywords | 著者キーワードとMeSH⽤語の統合(Qualifier含む場合はDescriptor/Qualifier 形式) |
| publication_types | 論⽂タイプ(RCT、Review、Case Reportsなど)のリスト |
コード詳細(コピペ⽤)
import xml.etree.ElementTree as ET
import json
def main(xml_string: str):
try:
root = ET.fromstring(xml_string)
except ET.ParseError:
return {"parsed_result": []}
articles = []
for article in root.findall('.//PubmedArticle'):
data = {}
# 1. pmid
pmid = article.find('.//PMID')
data['pmid'] = pmid.text if pmid is not None else ""
# 2. Title
title = article.find('.//ArticleTitle')
data['title'] = title.text if title is not None else ""
# 3.Abstract
abstract_texts = []
abstract_section = article.find('.//Abstract')
if abstract_section is not None:
for text_node in abstract_section.findall('AbstractText'):
label = text_node.get('Label')
content = text_node.text or ""
if label:
abstract_texts.append(f"[{label}] {content}")
else:
abstract_texts.append(content)
data['abstract'] = "\n".join(abstract_texts)
# 4. Authors & Affiliations
author_list = []
for author in article.findall('.//Author'):
last = author.find('LastName')
fore = author.find('ForeName')
if last is not None and fore is not None:
author_list.append(f"{fore.text} {last.text}")
elif last is not None:
author_list.append(last.text)
data['author'] = author_list
main_author_affil = ""
first_author = article.find('.//AuthorList/Author')
if first_author is not None:
aff = first_author.find('.//AffiliationInfo/Affiliation')
if aff is not None and aff.text:
main_author_affil = aff.text.strip()
data['main_author_affiliation'] = main_author_affil
# 5-1.Journal(in short) info
journal_inshort = article.find('.//ISOAbbreviation')
data['journal_inshort'] = journal_inshort.text if journal_inshort is not None else ""
# 5-2. Journal_info
journal = article.find('.//Journal/Title')
data['journal'] = journal.text if journal is not None else ""
# 6.Year
year = article.find('.//PubDate/Year')
if year is None:
year = article.find('.//DateCompleted/Year')
data['year'] = year.text if year is not None else ""
# 7.DOI
doi = ""
for eloc in article.findall('.//ELocationID'):
if eloc.get('EIdType') == 'doi':
doi = eloc.text
break
data['doi'] = doi
# 8.Keywords
keywords_set = set()
# A. 著者キーワード (KeywordList)
for kw in article.findall('.//Keyword'):
if kw.text:
keywords_set.add(kw.text.strip())
# B. MeSH用語 (MeshHeadingList)
for mesh_heading in article.findall('.//MeshHeading'):
descriptor = mesh_heading.find('DescriptorName')
if descriptor is not None and descriptor.text:
desc_text = descriptor.text.strip()
# Qualifier
qualifiers = mesh_heading.findall('QualifierName')
if len(qualifiers) > 0:
for q in qualifiers:
if q.text:
keywords_set.add(f"{desc_text}/{q.text.strip()}")
else:
keywords_set.add(desc_text)
# 9. Publication Types
pub_types = []
for pt in article.findall('.//PublicationTypeList/PublicationType'):
if pt.text:
pub_types.append(pt.text.strip())
data['publication_types'] = pub_types
# リストに戻してソート
data['MeSH_Keywords'] = sorted(list(keywords_set))
articles.append(data)
return {
"parsed_result" : articles
}
出力
| 出⼒名 | 型 | 説明 |
|---|---|---|
| parsed_result | array[object] | 1レコード1論⽂の辞書リスト |
4. まとめ
本記事で解説した内容
- E-Fetchによる論⽂詳細データの取得(XML形式)
- XMLレスポンスを構造化データへ変換するコード例
- 抽出される主要なフィールド(pmid、title、abstract、author、MeSH_Keywords、publication_typesなど)
次のステップ
Part 3では、ここで得た parsed_result を⼊⼒に、LLMでタイトル翻訳‧要約‧優先度判定を⾏います。イテレーションノードの並列処理やStructured Outputの活⽤法など、AI処理の中⼼部分を解説します。
シリーズ記事
- Part0: 全体像とPubMed API基礎
- Part 1: パラメータ抽出とE-Search編
- Part 2: E-Fetchとデータパース編
- Part 3(次回記事): AI処理‧データ整形編
- Part4: データ保存とGAS連携編

ヘルツレーベンでは、ライフサイエンス業界に特化したDX・自動化支援を提供しています。
PubMedや学術情報の自動収集をはじめ、Slack・Gmailなどを活用したナレッジ共有の仕組みまで、実務に直結するワークフローを設計・導入いたします。
提供サービスの例
- 製薬・医療機器業界での提案活動や調査業務の自動化支援
- アカデミアや研究者向けの文献レビュー・情報共有フローの最適化
- 医療従事者のキャリア開発を支援するリスキリングプログラム
👉 ご興味をお持ちの方はぜひお気軽にお問い合わせください。
お問い合わせフォームはこちら

監修者 株式会社ヘルツレーベン代表 木下 渉
株式会社ヘルツレーベン 代表取締役/医療・製薬・医療機器領域に特化したDXコンサルタント/
横浜市立大学大学院 ヘルスデータサイエンス研究科 修了
製薬・医療機器企業向けのデータ利活用支援、提案代行、営業戦略支援を中心に、医療従事者向けのデジタルスキル教育にも取り組む。AI・データ活用の専門家として、企業研修、プロジェクトPMO、生成AI導入支援など幅広く活動中





