Difyでつくる論⽂仕分けアプリ Part3: LLM処理‧データ保存編
| 目次 |
1. はじめに
本記事は、Difyのチャットワークフローを使って、PubMed論⽂の検索‧翻訳‧要約を⾃動化するシステムを構築するシリーズのPart 3です。
Part 2の復習: 前回の記事では、E-Fetchで論⽂詳細データを取得し、XMLをパースして構造化データを作るところまで解説しました。具体的には、以下のノードを実装しました。
- E-Fetch(XML形式で論⽂詳細データを取得)
- XMLパース(PythonでXMLを解析し、構造化データに変換)
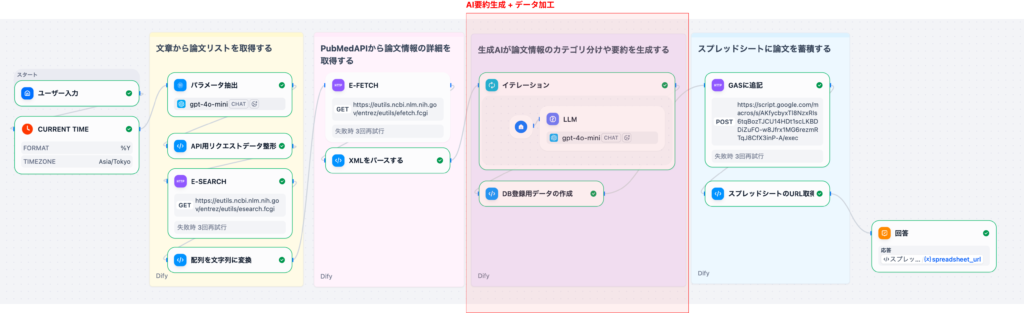
本記事(Part 3)では、取得した論⽂データに対してLLMで翻訳‧要約‧優先度判定を⾏い、CSV形式に整形する処理を詳しく解説します。この部分は、ワークフローの核⼼となるAI処理部分です。
シリーズ構成
- Part0: 全体像とPubMed API基礎
- Part 1: 検索・データ取得編
- Part 2: AI処理・データ整形編
- Part 3(本記事): LLM処理・データ保存編
- Part4: DifyとGAS連携で実現する可能性
2. ワークフローの位置付け
Part 2で取得したデータは、以下のような構造になっています。
E-Fetchを使った場合
{
"parsed_result": [
{
"pmid": "12345678",
"title": "Effect of Insulin Therapy in Type 2 Diabetes",
"abstract": "[Background] Type 2 diabetes...",
"author": ["John Smith", "Jane Doe"],
"journal": "Diabetes Research",
"year": "2024",
"doi": "10.1234/example",
"keywords": ["diabetes", "insulin", "therapy"]
}
]
}
本記事では、E-Fetchで取得した論⽂データに対して、以下の処理を⾏います。
- イテレーションで各論⽂を一つずつ処理
- LLMで各論⽂のタイトル翻訳・要約・優先度判定・研究領域抽出・対象抽出
- 元データとAI分析結果をマージしてCSV⽣成
3. 各ノードの詳細解説
3-1. イテレーション(Iteration)
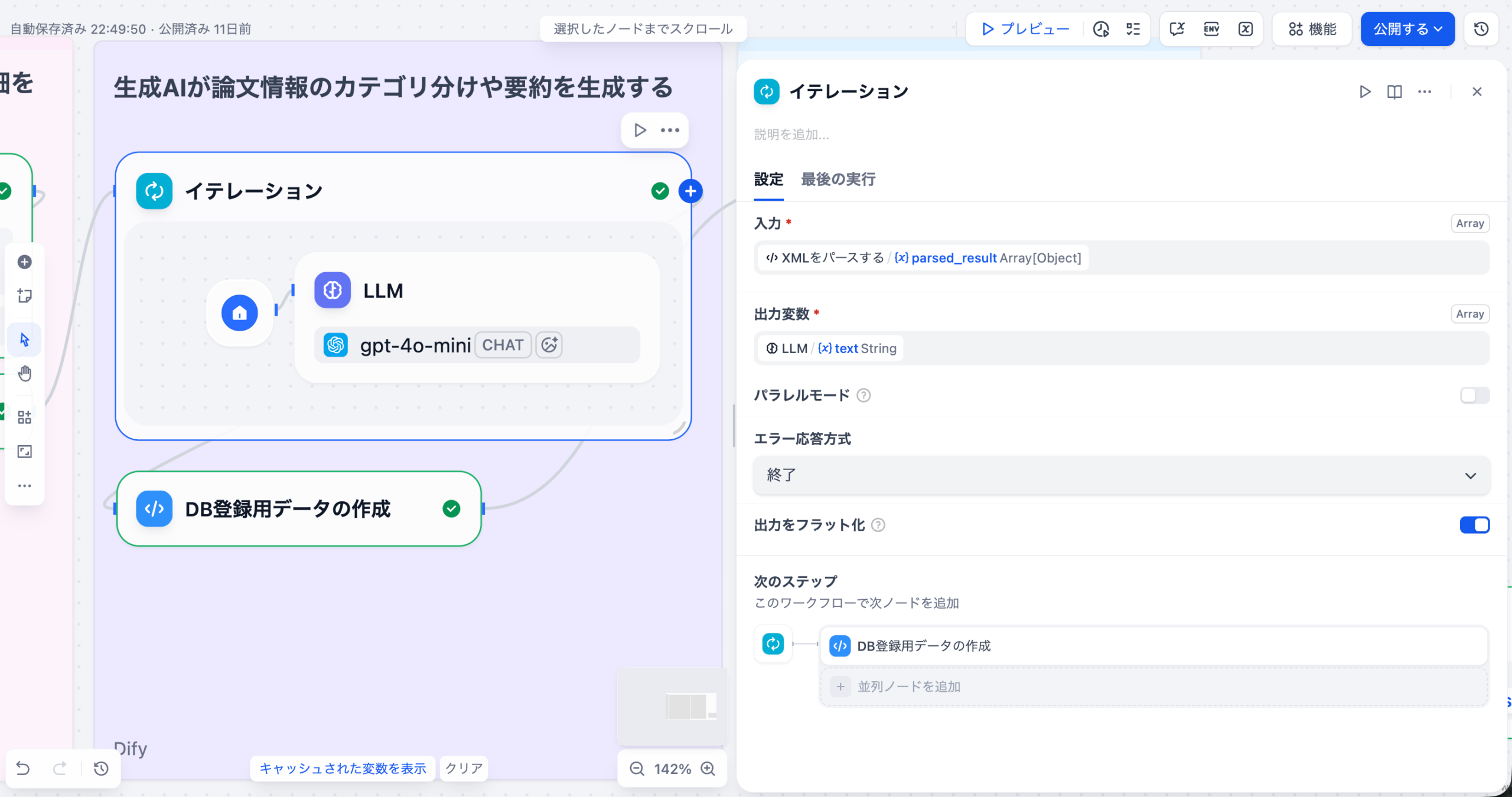
パースされた論⽂データの配列をループ処理し、各論⽂に対してLLMによる翻訳‧要約‧優先度判定を⾏うノードです。
イテレーションとは?
DifyのIterationノードは、リストの要素に対して同じ処理を繰り返すために使います。
たとえば、URLリストや論⽂リスト(論⽂1,論⽂2,論⽂3…)の⼀つ⼀つに同じAI処理を適⽤したいときに便利です。このノードは、プログラミングのfor⽂のように、リストのすべての項⽬を順に処理し、結果をまとめて出⼒します。
設定内容
| 項⽬ | 設定値 |
|---|---|
| 入力変数 | {{parsed_result}} |
| 出力変数 | {{text}} (後で説明するLLMノードを先に配置すると選択できるようになります) |
| エラーハンドリング | エラー時は終了 |
| 出力をフラット化 | true |
処理の流れ
- ⼊⼒: XMLパースノードから parsed_result (論⽂データの配列)を受け取る
- ループ: 各論⽂データを1件ずつ処理
- 出⼒: LLMの出⼒を配列として集約
3-2.LLM(イテレーション内側のノードです)
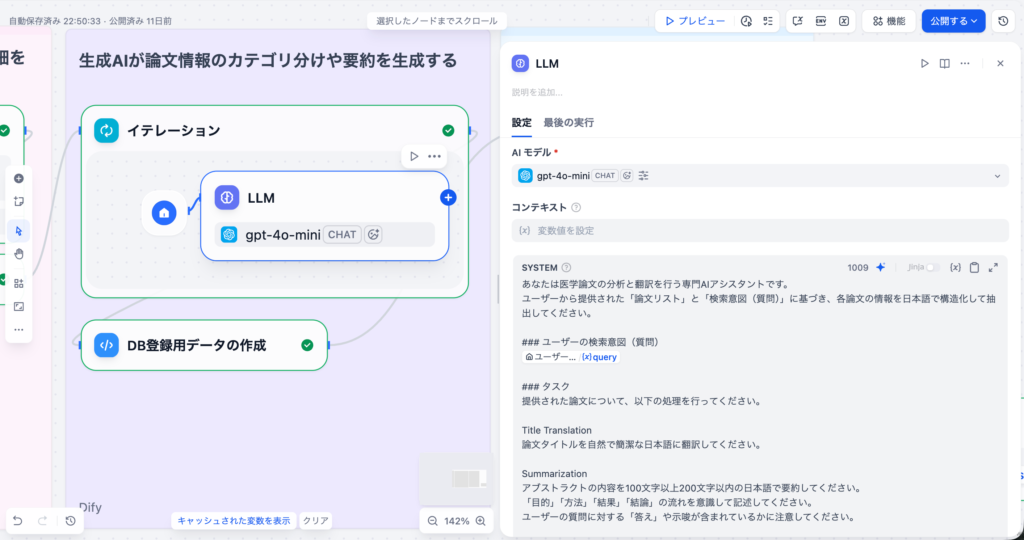
各論⽂に対して、タイトルの⽇本語翻訳、アブストラクトの要約、優先度判定を⾏うLLMノードです。イテレーション内に配置されており、各論⽂ごとに個別に処理されます。
イテレーションの中で、LLMノードを配置することで、実行するたびに各論文データに対して、一つずつLLMが実行されます。
プロンプトテンプレート
あなたは医学論文の分析と翻訳を行う専門AIアシスタントです。
ユーザーから提供された「論文リスト」と「検索意図(質問)」に基づき、各論文の情報を日本語で構造化して抽出してください。
### ユーザーの検索意図(質問)
{{#sys.query#}}
### タスク
提供された論文について、以下の処理を行ってください。
Title Translation
論文タイトルを自然で簡潔な日本語に翻訳してください。
Summarization
アブストラクトの内容を100文字以上200文字以内の日本語で要約してください。
「目的」「方法」「結果」「結論」の流れを意識して記述してください。
ユーザーの質問に対する「答え」や示唆が含まれているかに注意してください。
Priority Assessment
ユーザーの質問に対するその論文の重要度を3段階で判定してください。
HIGH: 質問の意図と高いレベルで一致し、かつRCT、メタアナリシス、システマティックレビューなど高いエビデンスレベル、または重要な新知見を含む。
MID: 質問と関連はあるが一部が周辺的、または観察研究・症例報告などエビデンスレベルが限定的。
LOW: 質問の意図と大きく異なる、対象が全く異なる(例:動物実験のみ)、または臨床的意義が小さい。
Research Area(研究領域)の抽出
論文のタイトル・アブストラクト・MeSH用語などから、主要な疾患領域・診療科・トピックを1〜3個程度、日本語で要約してください。
例:Oncology, Cardiovascular, Endocrinology, Psychiatry, Neurology, Infectious disease などを、日本語で「腫瘍学」「循環器」「内分泌」「精神科」「神経内科」「感染症」などと表現する。できるだけ専門領域名として通用する粒度で簡潔に記述してください。
Population(対象)の抽出
研究の対象となっている集団を日本語で要約してください。
年齢層(成人/高齢者/小児/新生児 など)
患者群(例:2型糖尿病患者、心不全患者、健常成人 など)
動物実験・細胞実験のみの場合はその旨を明記してください(例:「マウスモデル」「培養細胞」など)。
### 入力データ(論文リスト)
{{#item#}}
プロンプト設計のポイント
- 検索意図の活⽤: {{#sys.query#}} でユーザーの検索クエリを参照し、要約や優先度判定の基準として使⽤
- 構造化されたタスク: 5つの明確なタスク(翻訳、要約、優先度判定、研究領域抽出、対象抽出)を定義
- 優先度判定の基準: HIGH/MID/LOWの判定基準を明確に定義し、⼀貫性のある判定を実現
- 研究領域と対象の抽出: 論⽂の分類と検索に役⽴つ追加情報を抽出
構造化出⼒(Structured Output)
LLMの出⼒を構造化するため「構造化出⼒」機能を使⽤しています。
※構造化出力はAIのモデルによってサポートされていない場合があります。うまくいかない場合はバージョンを変えて試してみてください(gpt-4o-miniでは動作確認済み)。
| フィールド名 | 型 | 説明 |
|---|---|---|
| title_jp | string | 論⽂の⽇本語タイトル |
| summary | string | 要約(100〜200⽂字程度) |
| priority | string | 重要度(HIGH, MID, LOW) |
| research_area | array[string] | 研究領域(1〜3個程度、⽇本語) |
| population | string | 対象(年齢層‧患者群‧実験モデルなど) |
LLMによってこれらのラベルが自動的に付与されます。
出⼒例
各論⽂に対して以下のJSON形式で出⼒されます。
{
"title_jp": "糖尿病におけるインスリン療法の効果",
"summary": "本研究は、2型糖尿病患者におけるインスリン療法の有効性を検証した。無作為化比較試験により、インスリン療法群では血糖コントロールが有意に改善し、HbA1cが平均1.2%低下した。結論として、インスリン療法は2型糖尿病の効果的な治療選択肢であることが示された。",
"priority": "HIGH",
"research_area": ["内分泌", "糖尿病"],
"population": "2型糖尿病患者(成人)"
}
3-3. DB登録⽤データの作成(Codeノード)
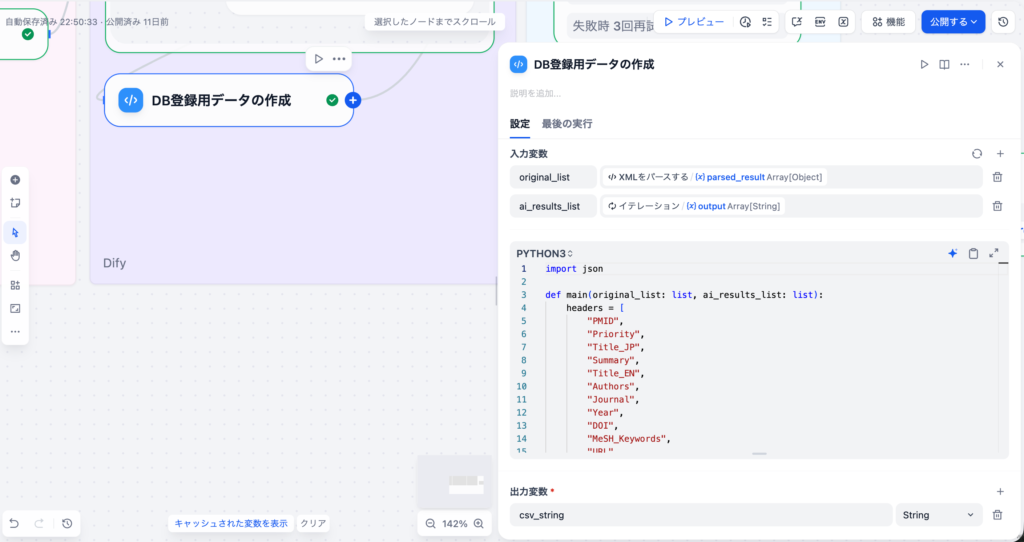
元の論⽂データ(XMLパース結果)とAI分析結果(LLM出⼒)をマージし、CSV形式に変換するノードです。
先ほど作成したLLMによる追加データとPubMed APIから取得したデータを統合して、一つの行データとして扱えるようにします。
⼊⼒変数
| 変数名 | ソース | 型 |
|---|---|---|
| original_list | XMLパースノード | array[object] |
| ai_results_list | イテレーションノード | array[string] |
CSVカラム構成
| カラム名 | 説明 | データソース |
|---|---|---|
| PMID | PubMed ID | 元データ |
| Priority | 重要度 | AI分析結果 |
| Title_JP | ⽇本語タイトル | AI分析結果 |
| Summary | 要約 | AI分析結果 |
| Title_EN | 英語タイトル | 元データ |
| Authors | 著者リスト | 元データ |
| Journal | 雑誌名 | 元データ |
| Year | 公開年 | 元データ |
| DOI | DOI | 元データ |
| MeSH_Keywords | MeSH⽤語とキーワード | 元データ |
| URL | PubMed URL | ⽣成( https://pubmed.ncbi.nlm.nih.gov/{pmid}/ ) |
| main_author_affiliation | 第⼀著者の所属機関 | 元データ |
| research_area | 研究領域 | AI分析結果 |
| publication_types | 論⽂タイプ | 元データ |
| population | 対象 | AI分析結果 |
コード詳細
以下はコピペでコードノードに貼り付けるだけで大丈夫です。
コードが動かない時には、「入力変数」「出力変数」の名前やデータ型が正しいかを確認してください。
import json
def main(original_list: list, ai_results_list: list):
headers = [
"PMID",
"Priority",
"Title_JP",
"Summary",
"Title_EN",
"Authors",
"Journal",
"Year",
"DOI",
"MeSH_Keywords",
"URL",
"main_author_affiliation",
"research_area",
"publication_types",
"population"
]
csv_rows = [",".join(['"' + h + '"' for h in headers])]
for i, original in enumerate(original_list):
ai_item = ai_results_list[i] if i < len(ai_results_list) else "{}"
ai_data = {}
try:
if isinstance(ai_item, dict):
ai_data = ai_item
else:
clean_json = str(ai_item).replace('```json', '').replace('```', '').strip()
ai_data = json.loads(clean_json)
except:
ai_data = {}
row_data = {}
pmid = original.get('pmid', '')
row_data["PMID"] = pmid
row_data["Title_EN"] = original.get('title', '')
auths = original.get('authors', original.get('author', []))
row_data["Authors"] = ", ".join(auths) if isinstance(auths, list) else str(auths)
row_data["Journal"] = original.get('journal', '')
row_data["Year"] = original.get('year', '')
row_data["DOI"] = original.get('doi', '')
row_data["main_author_affiliation"] = original.get('main_author_affiliation','')
row_data["publication_types"] = original.get('publication_types','')[0].replace('[','').replace(']','') if original.get('publication_types') else ''
kws = original.get('MeSH_Keywords', original.get('keyword', []))
row_data["MeSH_Keywords"] = ", ".join(kws) if isinstance(kws, list) else str(kws)
if pmid:
row_data["URL"] = f"<https://pubmed.ncbi.nlm.nih.gov/{pmid}/>"
else:
row_data["URL"] = ""
# LLM generated columns
row_data["Title_JP"] = ai_data.get('title_jp', '')
row_data["Summary"] = ai_data.get('summary', '')
row_data["Priority"] = ai_data.get('priority','')
research_area_list = ai_data.get('research_area', [])
if research_area_list and len(research_area_list) > 0:
row_data["research_area"] = research_area_list[0].replace('[','').replace(']','') if isinstance(research_area_list[0], str) else str(research_area_list[0])
else:
row_data["research_area"] = ''
row_data["population"] = ai_data.get('population','')
csv_row = []
for col in headers:
val = row_data.get(col, "")
val_escaped = str(val).replace('"', '""')
csv_row.append(f'"{val_escaped}"')
csv_rows.append(",".join(csv_row))
final_csv = "\\n".join(csv_rows)
return {
"csv_string": final_csv
}
処理の流れ
- ヘッダー⾏の⽣成: CSVのヘッダー⾏を作成
- ループ処理: 元データとAI分析結果を1件ずつ処理
- AI分析結果のパース: LLMの出⼒をJSONとして解析(エラーハンドリング付き)
- データマージ: 元データとAI分析結果を統合
- CSV⾏の⽣成: 各フィールドをエスケープ処理してCSV形式に変換
- URL⽣成: PMIDからPubMedのURLを⾃動⽣成
エスケープ処理の重要性
CSV形式では、フィールド内にカンマやダブルクォートが含まれる場合、適切にエスケープする必要があります。このコードでは、ダブルクォートを “” に変換することで、正しいCSV形式を保証しています。
出⼒
| 出⼒名 | 型 | 説明 |
|---|---|---|
| csv_string | string | CSV形式の⽂字列 |
出⼒例
以下のように出⼒することで、SpreadsheetやExcelで扱いやすいcsvの形式にしています。これによってSpreadsheetやExcelに連携する時のデータ変換処理が容易になります。
"PMID","Priority","Title_JP","Summary","Title_EN","Authors","Journal","Year","DOI","MeSH_Keywords","URL","main_author_affiliation","research_area","publication_types","population""12345678","HIGH","糖尿病におけるインスリン療法の効果","本研究は、2型糖尿病患者におけるインスリン療法の有効性を検証した。...","Effect of Insulin Therapy in Type 2 Diabetes","John Smith, Jane Doe","Diabetes Research","2024","10.1234/example","diabetes, insulin, therapy","<https://pubmed.ncbi.nlm.nih.gov/12345678/","University> of Tokyo","内分泌","Randomized Controlled Trial","2型糖尿病患者(成人)"
4. まとめ
本記事では、取得した論⽂データに対してLLMで翻訳‧要約‧優先度判定を⾏い、CSV形式に整形する処理を詳しく解説しました。
本記事で実現したこと
- イテレーションによる論⽂データのループ処理
- LLMによる各論⽂の翻訳‧要約‧優先度判定
- 元データとAI分析結果のマージ
- CSV形式への変換(エスケープ処理付き)
処理の流れの確認
- イテレーション: 論⽂データをループ処理
- LLM: 各論⽂に対して翻訳‧要約‧優先度判定‧研究領域抽出‧対象抽出
- DB登録⽤データの作成: 元データとAI分析結果をマージしてCSV⽣成
次のステップ
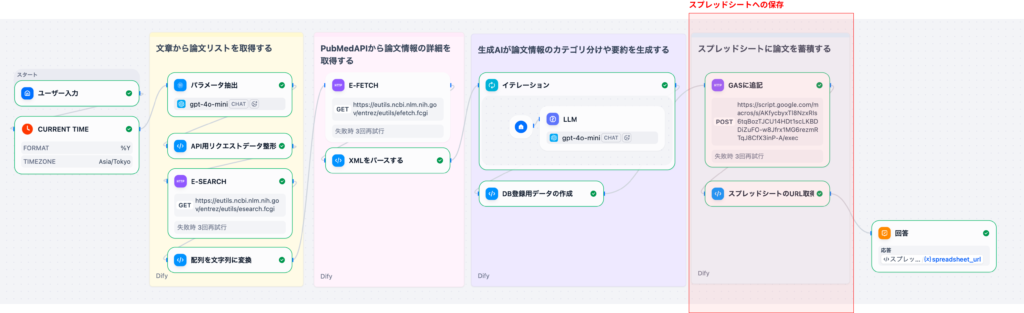
次回のPart 4では、⽣成したCSVデータをGoogle Apps Script(GAS)へ送信してスプレッドシートに保存する処理と、GAS連携で実現できる応⽤例を解説します。具体的には以下のテーマを扱います。
- CSV統合⽤の変数集約器
- GAS WebhookへのPOST送信
- レスポンスからスプレッドシートURLを取得するコード
- Dify × GAS連携の応⽤(通知、定期実⾏、他システムとの統合 等)
これらの処理により、ワークフローが完成し、ユーザーはスプレッドシートのURLを受け取って、保存された論⽂データを確認できるようになります。
シリーズ記事
- Part0: 全体像とPubMed API基礎
- Part 1: 検索・データ取得編
- Part 2: AI処理・データ整形編
- Part 3: LLM処理・データ保存編
- Part4(次回記事): DifyとGAS連携で実現する可能性

ヘルツレーベンでは、ライフサイエンス業界に特化したDX・自動化支援を提供しています。
PubMedや学術情報の自動収集をはじめ、Slack・Gmailなどを活用したナレッジ共有の仕組みまで、実務に直結するワークフローを設計・導入いたします。
提供サービスの例
- 製薬・医療機器業界での提案活動や調査業務の自動化支援
- アカデミアや研究者向けの文献レビュー・情報共有フローの最適化
- 医療従事者のキャリア開発を支援するリスキリングプログラム
👉 ご興味をお持ちの方はぜひお気軽にお問い合わせください。
お問い合わせフォームはこちら

監修者 株式会社ヘルツレーベン代表 木下 渉
株式会社ヘルツレーベン 代表取締役/医療・製薬・医療機器領域に特化したDXコンサルタント/
横浜市立大学大学院 ヘルスデータサイエンス研究科 修了
製薬・医療機器企業向けのデータ利活用支援、提案代行、営業戦略支援を中心に、医療従事者向けのデジタルスキル教育にも取り組む。AI・データ活用の専門家として、企業研修、プロジェクトPMO、生成AI導入支援など幅広く活動中





