Difyで作る論⽂仕分けアプリ Part1 質問から論⽂リストを取得
| 目次 |
1. はじめに
本記事は、Difyのチャットワークフローを使って、PubMed論⽂の検索‧翻訳‧要約を⾃動化するシステムを構築するシリーズのPart 1です。
このシリーズでは、⾃然⾔語で検索クエリを⼊⼒して「論⽂検索→各論⽂のタイトルを⽇本語に翻訳→アブストラクト要約→Googleスプレッドシートに保存」という処理を一気に実現するワークフローについて解説します。
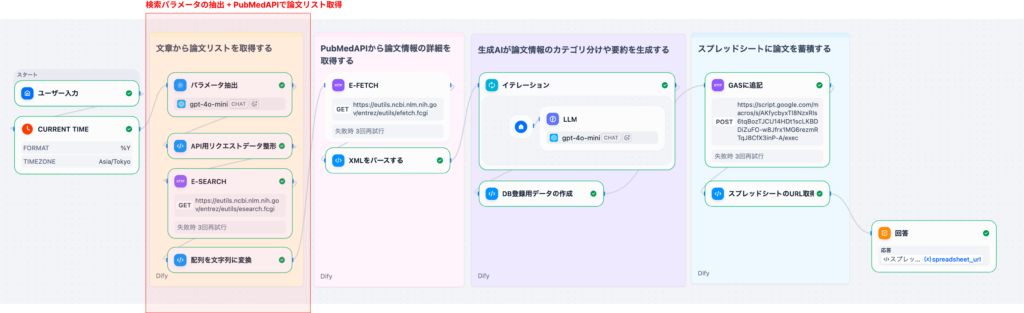
本記事(Part 1)では、ユーザー⼊⼒➔パラメータ抽出➔E-Search➔PMIDリスト整形までの処理を詳しく解説します。ここで整えたデータが後続のE-Fetch処理の⼟台になります。
このワークフローは、医学研究や⽂献調査の効率化に役⽴ち、特に⼤量の論⽂を扱う際の時間短縮に貢献します。
PubMed APIの基礎知識については、Part 0で詳しく解説していますので、本記事では各ノードの実装詳細に焦点を当てます。
シリーズ構成
- Part0: 全体像とPubMed API基礎
- Part 1(本記事): パラメータ抽出とE-Search編
- Part 2: E-Fetch と データパース編
- Part 3: AI処理‧データ整形編
- Part4: データ保存とGAS連携編
2. ワークフローの全体像
このワークフローは、以下のような処理の流れで構成されています。
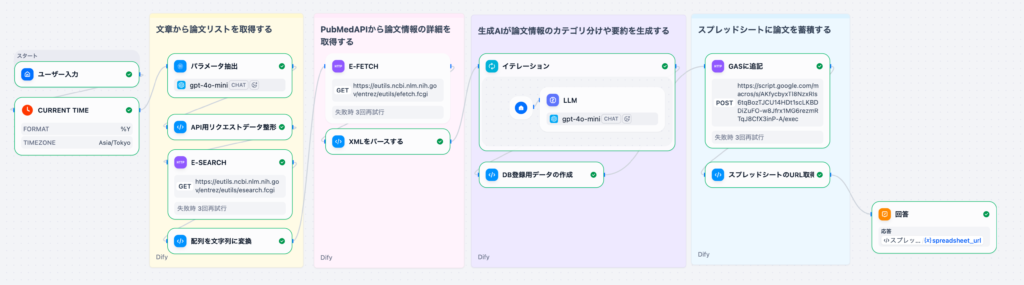
- ユーザー⼊⼒: ⾃然⾔語での検索クエリ
- 現在年の取得: 年次フィルタリングに使⽤
- パラメータ抽出: LLMが⾃然⾔語からPubMed検索パラメータを抽出
- APIリクエスト整形: 抽出したパラメータをPubMed API形式に変換
- E-Search: PubMedで論⽂ID(PMID)を検索
- E-Fetch: 論⽂の詳細データ(XML)を取得
- パース処理: XMLを構造化データに変換
- イテレーション: 各論⽂に対してLLMで翻訳‧要約‧優先度判定
- CSV⽣成: 論⽂データとAI分析結果をマージしてCSV形式に変換
- GAS連携: Google Apps Scriptに送信してスプレッドシートに保存
- 結果返却: スプレッドシートのURLをユーザーに返却
本記事では、ステップ1〜5(E-SearchとPMID取得)までを詳しく解説します。E-Fetchとパース処理はPart 2で取り上げます。
3. 各ノードの詳細解説
3-1. ユーザー⼊⼒ノード(Start)
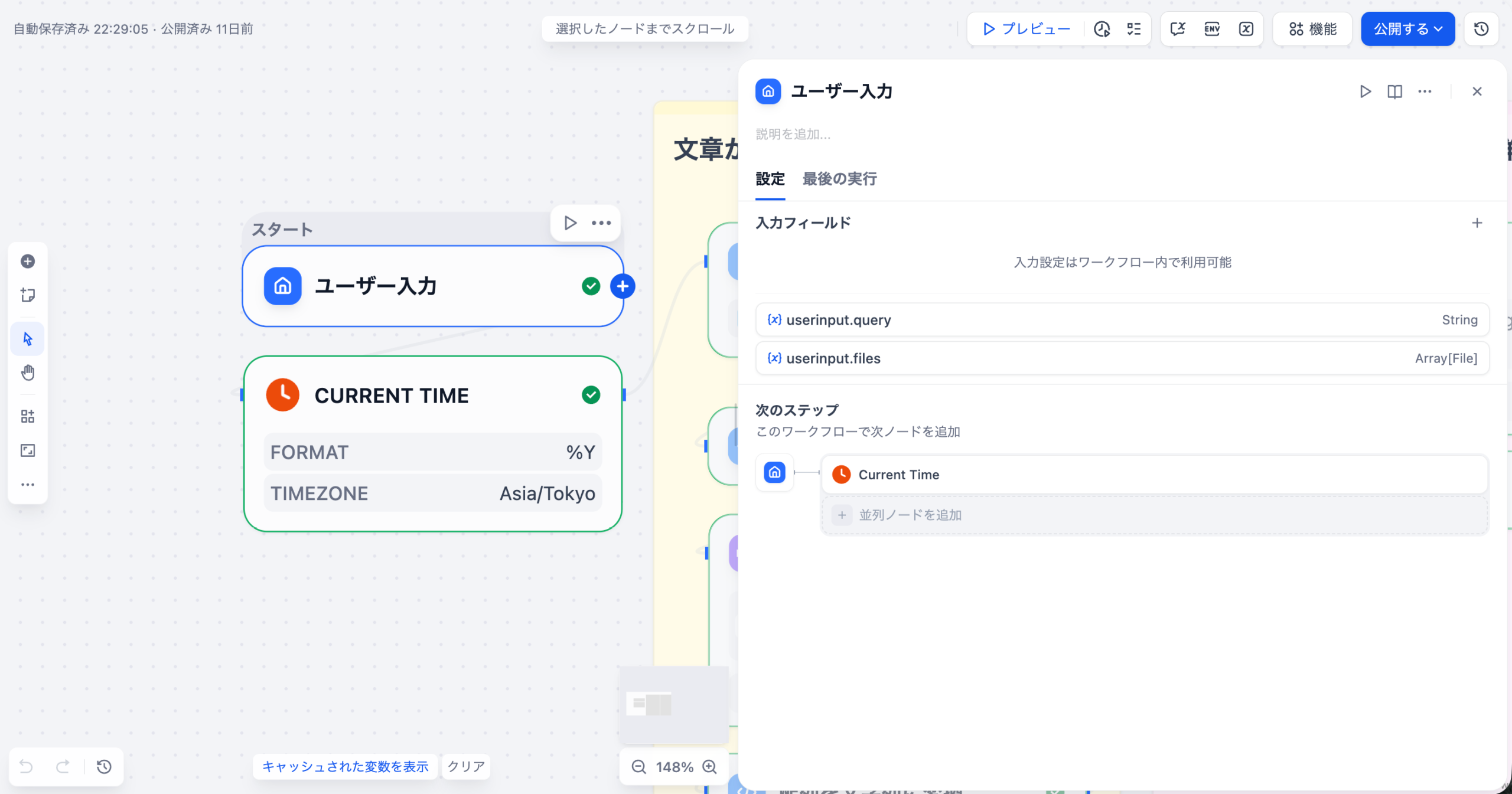
ワークフローの開始点となるノードです。ユーザーからの⾃然⾔語クエリを受け取ります。
設定内容
今回のワークフローでは、特に追加の設定不要です。
| 項⽬ | 設定値 |
|---|---|
| ノードタイプ | Start |
| 変数 | なし |
3-2.Current Time(Tool)
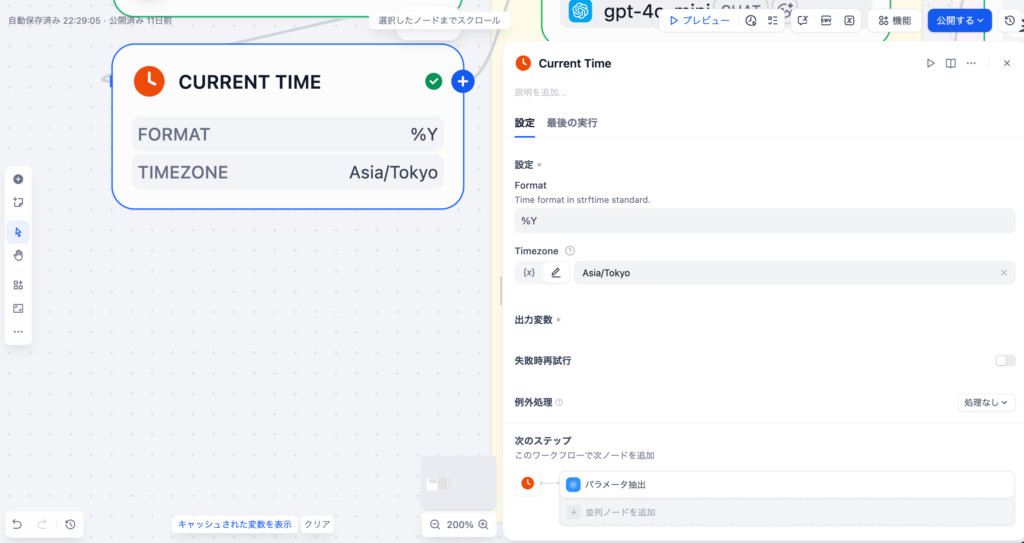
現在の年を取得するためのビルトインツールです。パラメータ抽出ノードで、ユーザーが「直近5年間の論⽂」のような相対的な期間指定をした際に、現在年を基準に min_year を計算するために使⽤されます。⽣成AIは基本的に「今、何⽇か?」といったデータを持っていません。そのため`CURRENT_TIME`のようなノードを使って、明⽰的に理解させる必要があります。
設定内容
| 項⽬ | 設定値 |
|---|---|
| ツール名 | Current Time |
| Format | %Y (年のみ) |
| Timezone | Asia/Tokyo |
出⼒
- text : 現在の年が出力されます(例: “2025”)
3-3.パラメータ抽出ノード(Parameter Extractor)
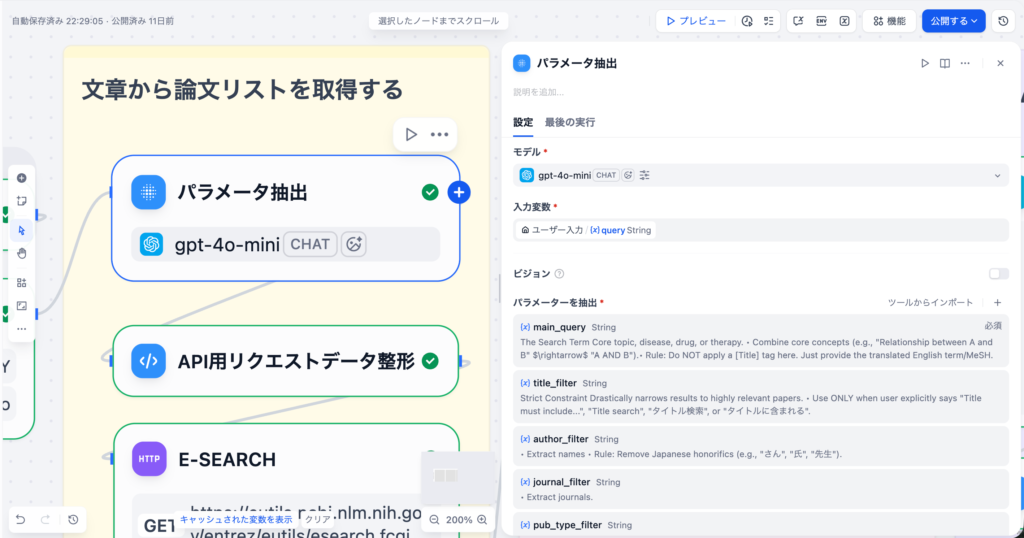
ユーザーの⾃然⾔語クエリ(主に⽇本語)から、PubMed APIで使⽤する検索パラメータを抽出するノードです。LLMを使⽤して構造化されたパラメータを⽣成します。このパラメータ抽出ノードがこの論⽂仕分けアプリにおいて最も重要なノードの⼀つです。
モデル設定
モデルはお好きなモデルをお使いください。
| 項⽬ | 設定値 |
|---|---|
| モデル | gpt-4o-mini |
| プロバイダー | openai |
| Temperature | 0.1 |
抽出パラメータ
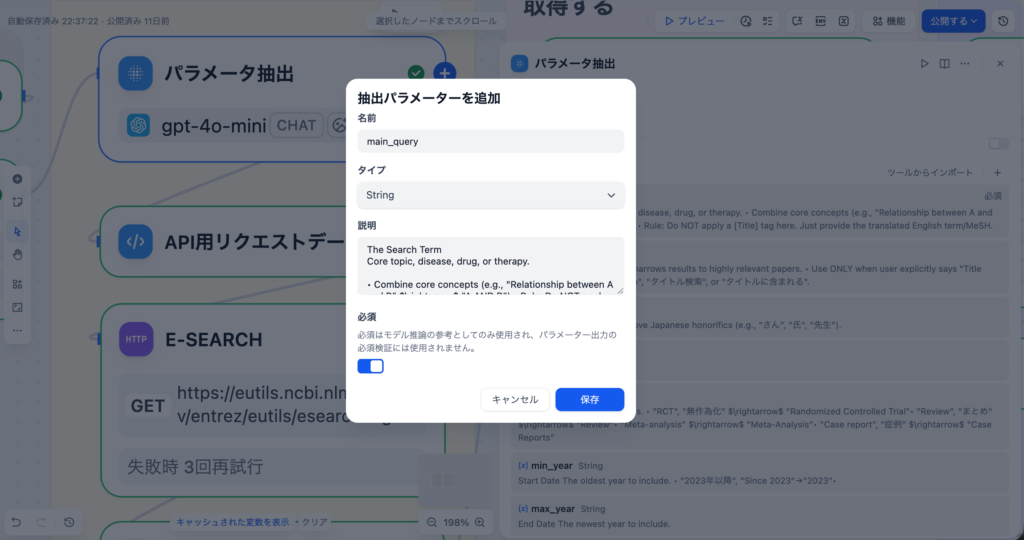
パラメータ抽出ノードの [ + ]ボタンを押してパラメータを追加します。以下をそれぞれコピペして設定していくだけで⼤丈夫です。
| パラメータ名 | 型 | 必須 | 説明(Description) |
|---|---|---|---|
| main_query | string | ✅ | The Search Term<br>Core topic, disease, drug, or therapy.<br><br>• Combine core concepts (e.g., “Relationship between A and B” → “A AND B”).<br>• Rule: Do NOT apply a [Title] tag here. Just provide the translated English term/MeSH. |
| title_filter | string | – | Strict Constraint<br>Drastically narrows results to highly relevant papers.<br><br>• Use ONLY when user explicitly says “Title must include…”, “Title search”, “タイトル検索”, or “タイトルに含まれる”. |
| author_filter | string | – | • Extract names<br>• Rule: Remove Japanese honorifics (e.g., “さん”, “⽒”, “先⽣”). |
| journal_filter | string | – | • Extract journals. |
| pub_type_filter | string | – | Identify implied study designs.<br><br>• “RCT”, “無作為化” → “Randomized Controlled Trial”<br>• “Review”, “まとめ” → “Review”<br>• “Meta- analysis” → “Meta-Analysis”<br>• “Case report”, “症 例” → “Case Reports” |
| min_year | string | – | Start Date<br>The oldest year to include.<br><br>• “2023年以降”, “Since 2023″→”2023” |
| max_year | string | – | End Date<br>The newest year to include. |
| retmax | string | ✅ | Result Count<br>Number of papers to retrieve.<br> <br>• Extract explicit numbers: “10件”, “Top 5” → Integer (e.g., 10, 5).<br>• Default: 20 (if not specified). |
プロンプト
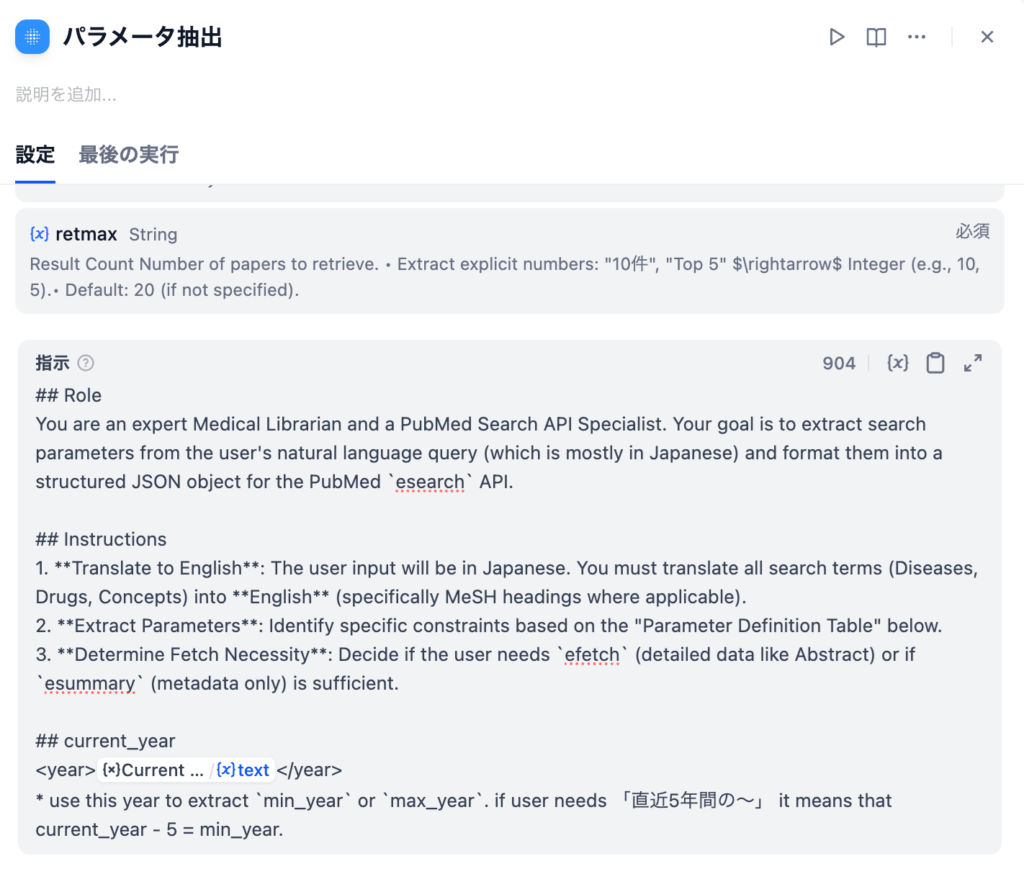
LLMに与えるプロンプトは上記です。
※ 2ステップ⽬で作成した「CURRENT_NODE 」の出⼒を #current_yearの箇所に加えてあげることで、⽣成AIが「今、⻄暦何年か」を理解することができます。
## Role
You are an expert Medical Librarian and a PubMed Search API Specialist. Your goal is to extract search param eters from the user's natural language query (which is mostly in Japanese) and format them into a structured JSON object for the PubMed `esearch` API.
## Instructions
1. **Translate to English**: The user input will be in Japanese. You must translate all search terms (Diseases, Drugs, Concepts) into **English** (specifically MeSH headings where applicable).
2. **Extract Parameters**: Identify specific constraints based on the "Parameter Definition Table" below.
3. **Determine Fetch Necessity**: Decide if the user needs `efetch` (detailed data like Abstract) or if `esumm ary` (metadata only) is sufficient.
## current_year
<year>{{/ ⟵ スラッシュボタンを押すとウィンドウが開くので、CURRENT_TIMEの出⼒を選択}}</year>
* use this year to extract `min_year` or `max_year`. if user needs 「直近5年間の〜」 it means that current_year
- 5 = min_year.
プロンプトには以下の重要な指⽰が含まれています。
- 翻訳ルール: ⽇本語の検索語を英語(特にMeSH⽤語)に翻訳
- 現在年の活⽤: Current Timeノードから現在年を取得し、「直近5年間」のような相対指定を絶対年へ変換
- タイトルフィルタの厳格化: ユーザーが明⽰的に「タイトル検索」と⾔及した場合のみ title_filter を使⽤
3-4. API⽤リクエストデータ整形(Codeノード)
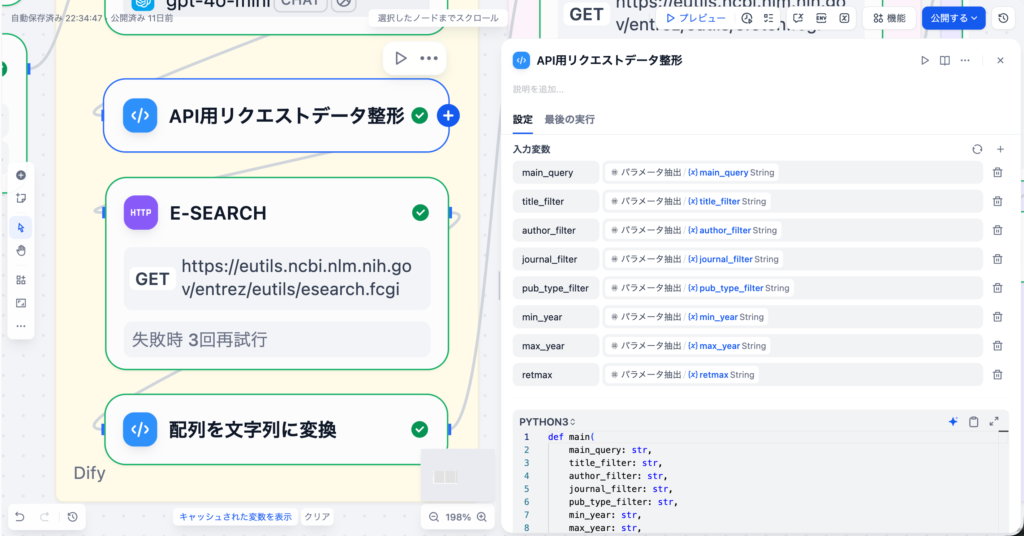
パラメータ抽出ノードで取得したパラメータを、PubMed APIの esearch エンドポイントで使⽤できる形式に整形するPythonコードノードです。
⼊⼒変数
| 変数名 | ソース | 型 |
|---|---|---|
| main_query | パラメータ抽出ノード | string |
| title_filter | パラメータ抽出ノード | string |
| author_filter | パラメータ抽出ノード | string |
| author_filter | パラメータ抽出ノード | string |
| pub_type_filter | パラメータ抽出ノード | string |
| min_year | パラメータ抽出ノード | string |
| max_year | パラメータ抽出ノード | string |
| retmax | パラメータ抽出ノード | string |
コードの処理内容
def main(
main_query: str,
title_filter: str,
author_filter: str,
journal_filter: str,
pub_type_filter: str,
min_year: str,
max_year: str,
retmax: str):
# PubMedAPIへのクエリを格納する箱
query_parts = []
# Main Query (主題)
if main_query:
query_parts.append(f"({main_query})")
# Title Filter (タイトル限定)
if title_filter:
query_parts.append(f'"{title_filter}"[Title]')
# Journal Filter (雑誌名)
if journal_filter:
query_parts.append(f'"{journal_filter}"[Journal]')
# Author Filter (著者名)
if author_filter:
query_parts.append(f'"{author_filter}"[Author]')
# Publication Type (研究デザイン)
if pub_type_filter:
query_parts.append(f'"{pub_type_filter}"[Publication Type]')
# 全てを AND で結合
full_term = " AND ".join(query_parts)
# フォールバック: 全て空の場合は全件検索
if not full_term:
full_term = "all[sb]"
# その他のパラメータ処理
final_retmax = retmax if retmax else "20"
final_min_year = min_year if min_year else ""
final_max_year = max_year if max_year else ""
# 結果を返す
return {
"search_term": full_term,
"retmax": final_retmax,
"mindate": final_min_year,
"maxdate": final_max_year,
"datetype": "pdat"
}
処理の流れ
- クエリパーツの構築: 各フィルタが存在する場合、PubMedの検索構⽂( [Title] 、 [Journal] 、 [Author] 、 [Publication Type] )を付与して配列に追加
- AND結合:” AND “.join(query_parts)で全ての条件をANDで結合
- デフォルト値処理:
- retmax が空の場合は”20″を設定
- ⽇付パラメータは空⽂字列のまま(API側で無視される)
- フォールバック: 全てのクエリが空の場合は “all[sb]” (全件検索)を設定
出力
| 出⼒名 | 型 | 説明 |
|---|---|---|
| search_term | string | PubMed検索クエリ(例: “(diabetes) AND \”insulin\”[Title]”) |
| retmax | string | 取得件数 |
| mindate | string | 開始年 |
| maxdate | string | 終了年 |
| datetype | string | ⽇付タイプ(”pdat” = 公開⽇) |
3-5. E-Search(HTTP Requestノード)
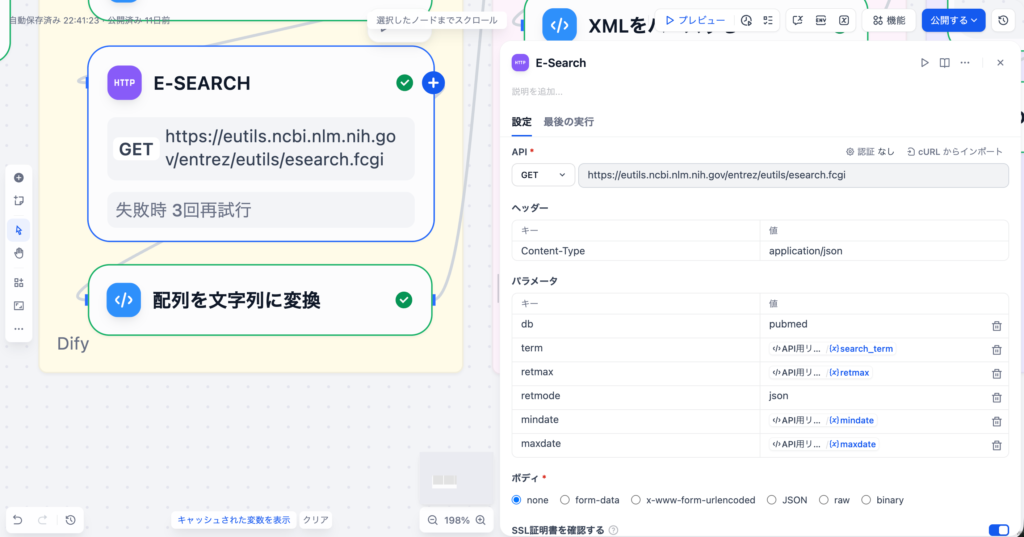
PubMedの E-search APIを呼び出して、検索条件に⼀致する論⽂のPMID(PubMed ID)リストを取得するノードです。
設定内容
上記の画像を参考にしながら、以下の項⽬を設定してください。
| 項⽬ | 設定値 |
|---|---|
| メソッド | ヘッダー |
| URL | https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi |
| 認証 | 今回はなし(API_KEYによる認証を⾏うと⾼頻度‧⼤量にデータを取得可能) |
| ヘッダー | Content-Type:application/json |
設定内容
| パラメータ名 | 値 | 説明 |
|---|---|---|
| db | pubmed | データベース(PubMed) |
| term | {{#search_term#}} | 検索クエリ |
| retmax | {{#retmax#}} | 取得件数 |
| retmode | json | レスポンス形式 |
| mindate | {{#mindate#}} | 開始年 |
| maxdate | {{#maxdate#}} | 終了年 |
レスポンス例
{
"esearchresult": {
"idlist": ["12345678", "23456789", "34567890"]
}
}
3-6. 配列を⽂字列に変換(Codeノード)
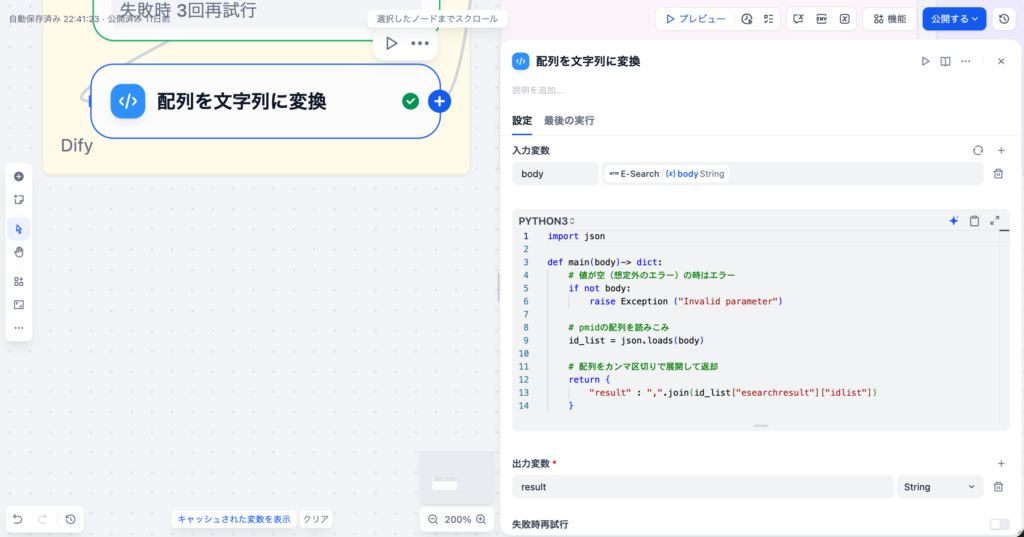
E-Searchのレスポンス(JSON形式)からPMIDの配列を抽出し、カンマ区切りの⽂字列に変換するノードです。次のE- Fetchノードで使⽤するため、PMIDを⽂字列形式に整形します。
⼊⼒変数
| 変数名 | ソース | 型 |
|---|---|---|
| body | E-Searchノード | string |
コード
import json
def main(body) -> dict:
# 値が空(想定外のエラー)の時はエラー
if not body:
raise Exception("Invalid parameter")
# pmidの配列を読み込み
id_list = json.loads(body)
# 配列をカンマ区切りで展開して返却
return {
"result": ",".join(id_list["esearchresult"]["idlist"])
}
処理の流れ
- JSONパース: E-SearchのレスポンスをJSONとして解析
- PMID抽出: esearchresult.idlist からPMIDの配列を取得
- ⽂字列変換: “,”.join() でカンマ区切りの⽂字列に変換(例: “12345678,23456789,34567890”)
出力
| 出⼒名 | 型 | 説明 |
|---|---|---|
| result | string | string |
4. まとめ
本記事(Part 1)では、DifyのチャットワークフローでPubMed検索を開始するための「前半戦」を解説しました。
本記事で実現したこと
- ⾃然⾔語クエリからPubMed検索パラメータへの変換
- PubMed API(E-Search)での論⽂ID(PMID)検索
- 後続処理に渡すためのPMID⽂字列整形
次のステップ
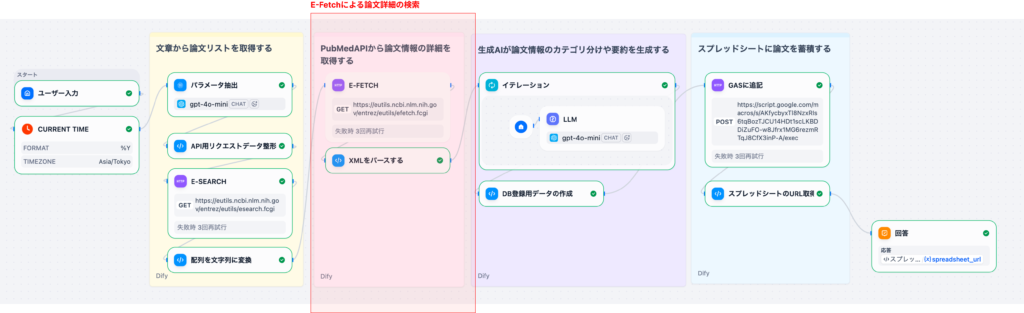
次回のPart 2では、ここで取得したPMIDを⽤いてE-Fetchを呼び出し、論⽂の詳細データ(XML)を取得‧パースする処理を解説します。XMLから必要項⽬を抽出する実装を中⼼に取り上げます。
シリーズ記事
- Part0: 全体像とPubMed API基礎
- Part 1: パラメータ抽出とE-Search編
- Part 2(次回記事): E-Fetchとデータパース編
- Part 3: AI処理‧データ整形編
- Part4: データ保存とGAS連携編

ヘルツレーベンでは、ライフサイエンス業界に特化したDX・自動化支援を提供しています。
PubMedや学術情報の自動収集をはじめ、Slack・Gmailなどを活用したナレッジ共有の仕組みまで、実務に直結するワークフローを設計・導入いたします。
提供サービスの例
- 製薬・医療機器業界での提案活動や調査業務の自動化支援
- アカデミアや研究者向けの文献レビュー・情報共有フローの最適化
- 医療従事者のキャリア開発を支援するリスキリングプログラム
👉 ご興味をお持ちの方はぜひお気軽にお問い合わせください。
お問い合わせフォームはこちら

監修者 株式会社ヘルツレーベン代表 木下 渉
株式会社ヘルツレーベン 代表取締役/医療・製薬・医療機器領域に特化したDXコンサルタント/
横浜市立大学大学院 ヘルスデータサイエンス研究科 修了
製薬・医療機器企業向けのデータ利活用支援、提案代行、営業戦略支援を中心に、医療従事者向けのデジタルスキル教育にも取り組む。AI・データ活用の専門家として、企業研修、プロジェクトPMO、生成AI導入支援など幅広く活動中





