Difyで作る医学論⽂仕分けアプリ: 全体像とPubMedAPI基礎
| 目次 |
1. はじめに
本シリーズでは、Difyのチャットワークフローを使⽤して、PubMed論⽂の検索‧翻訳‧要約を⾃動化するシステムの構築⽅法を解説します。
⾃然⾔語で検索クエリを⼊⼒すると、論⽂を検索し、各論⽂のタイトルを⽇本語に翻訳、アブストラクトを要約し、優先度を判定した上で、Googleスプレッドシートに保存するまでの⼀連の流れを実現します。
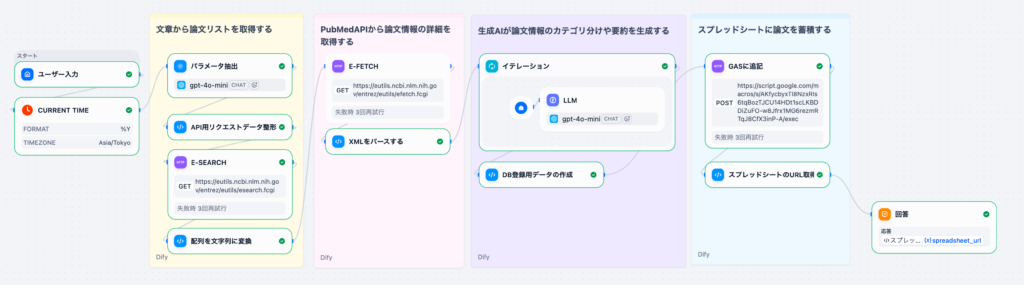
このワークフローは、医学研究や⽂献調査の効率化に役⽴ち、特に⼤量の論⽂を扱う際の時間短縮に貢献します。
1-1. 完成イメージ
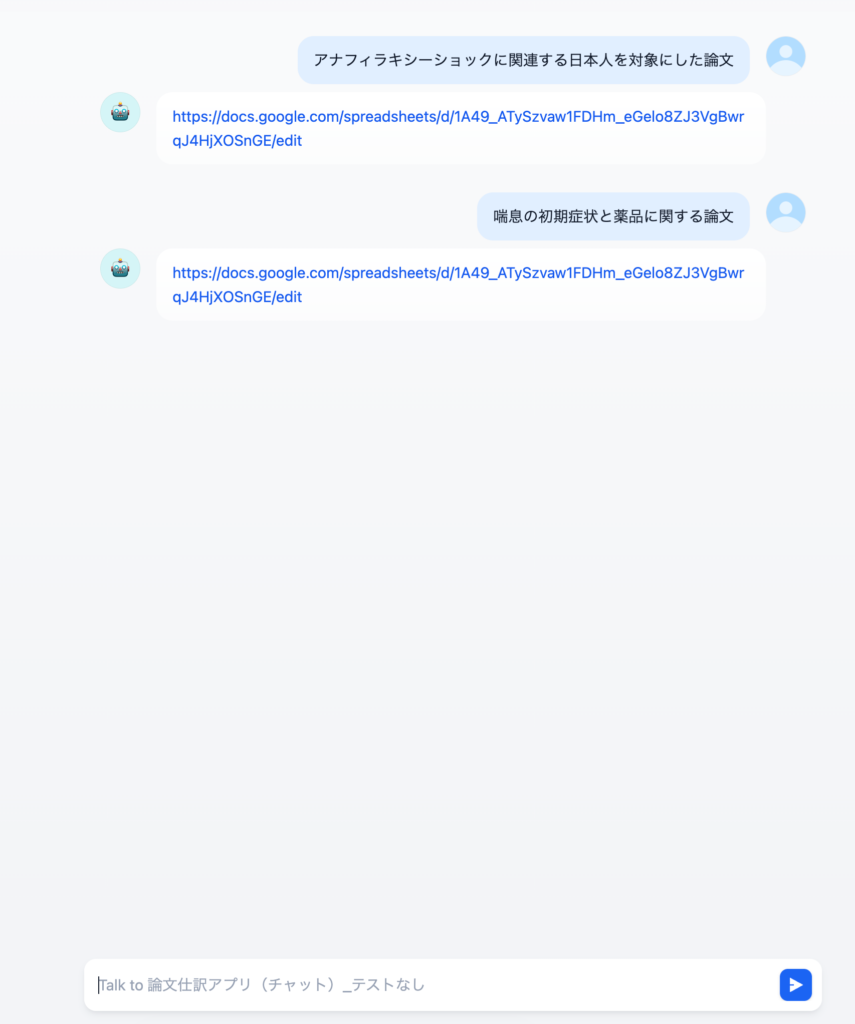

本記事(Part 0)では、ワークフローの全体像とPubMed APIの基礎知識を解説します。これらを理解することで、以降のPart 1〜Part 4で解説する各ノードの実装がより深く理解できるようになります。
シリーズ構成
- Part0(本記事): 全体像とPubMed API基礎
- Part 1: パラメータ抽出とE-Search編
- Part 2: E-Fetchとデータパース編
- Part 3: AI処理‧データ整形編
- Part4: データ保存とGAS連携編
2. ワークフローの全体像
このワークフローは、以下の5つの主要なステップで構成されています。
2-1. ステップ1: 検索パラメータの抽出(Part 1)
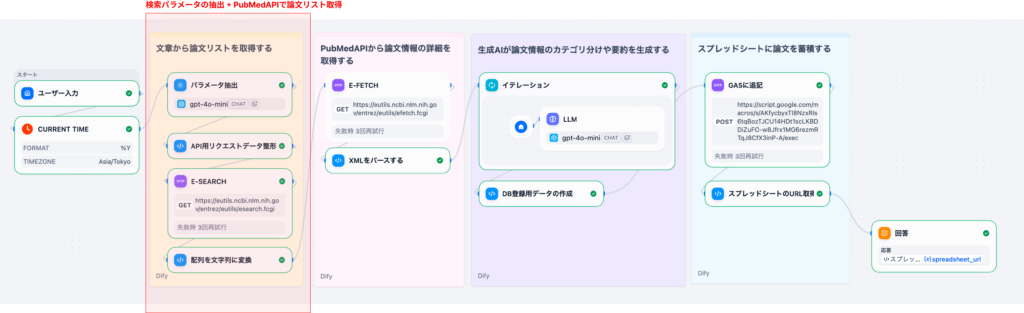
ユーザーが⾃然⾔語で⼊⼒した検索クエリ(例: 「糖尿病のインスリン療法に関する2020年以降のRCT」)を、PubMed APIで使⽤できる検索パラメータに変換します。
⼊⼒: ⾃然⾔語クエリ(⽇本語)
- 処理: LLMによるパラメータ抽出
- 出⼒: 構造化された検索パラメータ( main_query , title_filter , author_filter 等)
2-2. ステップ2: E-Fetchとデータパース(Part 2)
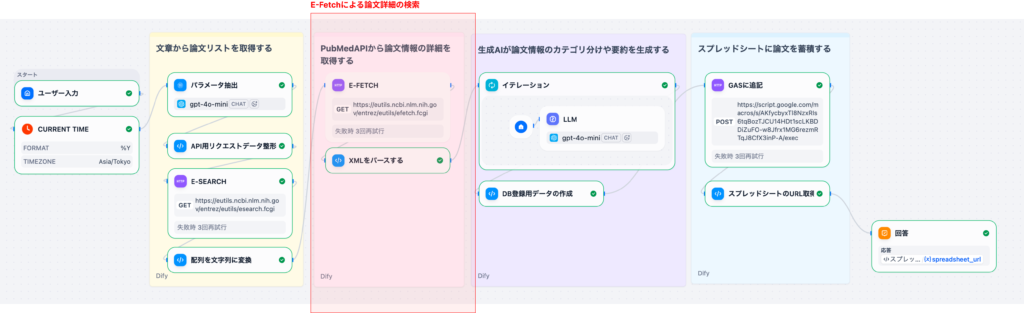
Part 1で⽣成したPMIDリストをもとに、⽤途に応じてE-Fetchまで論⽂詳細データを取得します。
- 論⽂詳細取得: E-Fetchによるデータ取得
- E-Fetch: XMLレスポンスを取得
- XML/JSONパース: LLMが扱いやすいPython dict/listへ整形
2-3. ステップ3: AIによる要約⽣成(Part 3)
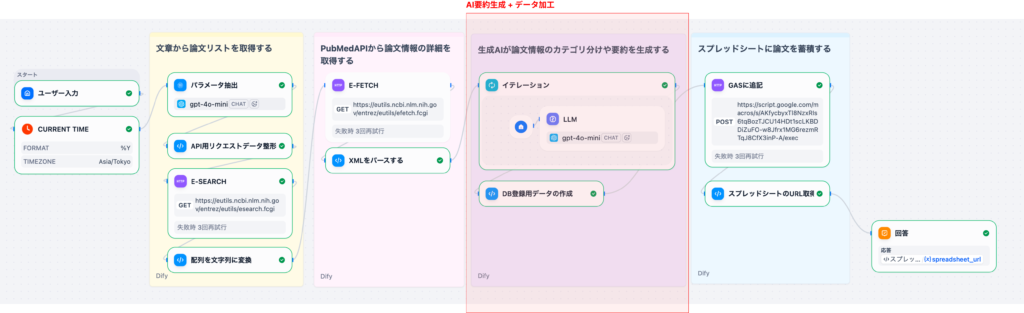
取得した論⽂データに対して、LLMを使⽤して以下の処理を⾏います。
- タイトル翻訳: 英語のタイトルを⾃然な⽇本語に翻訳
- 要約⽣成: アブストラクトを100〜200⽂字の⽇本語で要約
- 優先度判定: ユーザーの検索意図に基づいて、各論⽂の重要度をHIGH/MID/LOWで判定
- データマージ: 元データとAI分析結果を統合
- CSV⽣成: スプレッドシート保存⽤のCSV形式に変換
2-4. ステップ4: スプレッドシートへの保存(Part 4)
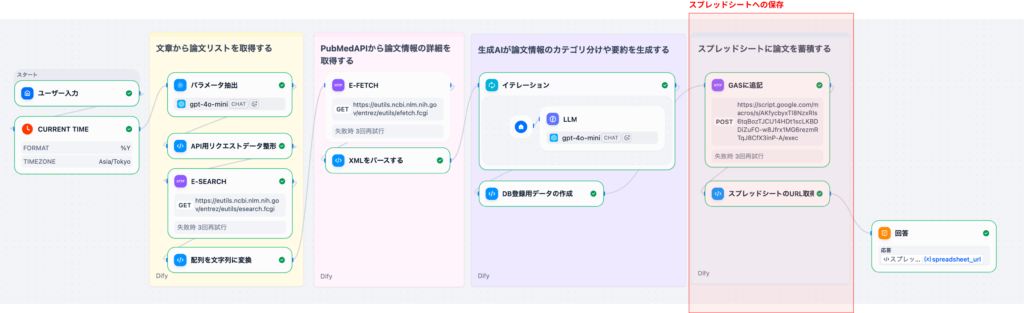
⽣成したCSVデータをGoogle Apps Script(GAS)経由でGoogleスプレッドシートに保存します。
- GAS連携: CSVデータをGASのWebhookエンドポイントに送信
- スプレッドシート保存: GASがCSVをパースしてスプレッドシートに追記
- 結果返却: スプレッドシートのURLをユーザーに返却
2-5.全体のデータフロー
ユーザー⼊⼒(⾃然⾔語) ↓ パラメータ抽出(LLM) ↓ E-Search(PMIDリスト取得) ↓ E-Fetch(詳細データ取得) ↓ XML/JSONパース ↓ イテレーション + LLM(翻訳‧要約‧優先度判定) ↓ CSV⽣成 ↓ GAS連携(スプレッドシート保存) ↓ 結果返却(URL)
3. PubMed API基礎知識
ワークフローの解説に映る前に、part0となる本記事では、PubMed APIについて解説します。
3-1. PubMed APIとは

PubMed APIは、⽶国国⽴医学図書館(NLM)が提供する⽣物医学分野の⽂献データベース「PubMed」にプログラムからアクセスするためのインターフェースです。正式名称は「Entrez Programming Utilities(E-utilities)」または「E-Utils」と呼ばれます。
このAPIを使⽤することで、プログラムからPubMedのデータを検索し、論⽂情報を⾃動的に取得‧処理することが可能になります。
3-2. 基本的な使⽤フロー
PubMed APIを使⽤する際は、以下の3つのステップを順番に実⾏する必要があります。
ステップ1: E-Search – 論⽂のリスト(PMID)を取得
まず、E-Searchを使⽤して、特定のキーワードや検索条件に合致する論⽂のPubMed ID(PMID)のリストを取得します。
重要なポイント: E-Searchは論⽂の詳細情報を返すのではなく、検索結果に該当する論⽂のPMID(識別番号)のリストのみを返します。このリストを取得することが、後続の処理の第⼀歩となります。
使用例:
https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&term=cancer
このリクエストにより、キーワード「cancer」に該当する論⽂のPMIDリストを取得できます。
ステップ2: E-SummaryまたはE-Fetch – 詳細情報を取得
E-Searchで取得したPMIDリストを基に、E-SummaryまたはE-Fetchを使⽤して各論⽂の詳細情報を取得します。
3-3. E-SummaryとE-Fetchの使い分け
E-Summary: 軽量な概要情報の取得
取得できる情報:
- タイトル
- 著者名
- 掲載誌名
- 出版年
- 基本的なメタデータ
特徴:
- データ量が少なく、処理が⾼速
- 多数の論⽂の概要を⼀括で把握するのに適している
- アブストラクト(要旨)は含まれない
使⽤例:
https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esummary.fcgi?db=pubmed&id=12345678
E-Fetch: 詳細情報の取得
E-Fetchの⽅が詳細取得が可能なため、今回のワークフローではこちらをメインに使⽤します。
取得できる情報:
- E-Summaryで取得できるすべての情報
- アブストラクト(要旨)
- MeSH⽤語(医学主題⾒出し)より詳細なメタデータ
- 全⽂へのリンク(利⽤可能な場合)
特徴:
- アブストラクトや詳細な情報が必要な場合に使⽤
- データが多いので必要最⼩限の論⽂に対して使⽤すると効率的
使⽤例:
<https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi?db=pubmed&id=12345678&retmode=xml>
使い分けの指針
- まずE-Summaryで概要を確認: 多数の論⽂を処理する場合や、タイトルや著者などの基本情報だけで⼗分な場合は、E-Summaryを使⽤します。これにより、処理速度を向上させることができます。
- 必要に応じてE-Fetchで詳細を取得: アブストラクトやMeSH⽤語など、より詳細な情報が必要な場合のみ、E-Fetchを使⽤します。E- Fetchはデータ量が多いため、必要な論⽂に対してのみ使⽤することを推奨します。
- 効率的な処理フロー:
- E-SearchでPMIDリストを取得
- E-Summaryで全論⽂の概要を確認
- 必要な論⽂のみを選別
- 選別した論⽂に対してE-Fetchで詳細情報を取得
今回作成した論⽂仕分けアプリは、最終的にスプレッドシートに論⽂を蓄積していくことも⽬標の⼀つです。そのため、E-Summaryは使わずにE-Fetchを活⽤したフローとなっています。ただE-Summaryも使えるようなフローを作成したので興味がある⽅は、後続の記事を読んで試してみてください。
3-4. 注意点
レート制限
- 1秒間に3回以上のリクエストを⾏わないようにする必要があります
- 過度なリクエストを⾏うと、アクセスが制限される可能性があります
- ⼤量のデータを取得する場合は、適切な間隔を設けてリクエストを⾏います
APIキー
- APIキーを取得することで、秒あたりのアクセス上限を増やすことが可能です
- APIキーはNCBIのアカウントから取得できます
利⽤規約
- NCBIの利⽤規約を遵守する必要があります
- 商⽤利⽤や⼤量のデータ取得を⾏う場合は、特に注意が必要です
3-5. PubMed APIのまとめ
PubMed APIを使⽤する際の基本的な流れは以下の通りです:
- E-Search: 検索条件に基づいてPMIDリストを取得(必須の第⼀歩)
- E-Summary: 基本的な情報を⾼速に取得(概要把握に適している)
- E-Fetch: 詳細な情報を取得(アブストラクトなどが必要な場合のみ)
この3つのAPIを適切に組み合わせることで、効率的にPubMedから論⽂情報を取得し、研究や業務の効率化を図ることができます。
本ブログシリーズで解説するワークフローでは、E-Summaryは使⽤しませんが、E-Fetchと同様の⽅法で情報が取得できるため、興味のある⽅は試してみて下さい。
4. このワークフローで実現すること
このワークフローを構築することで、以下のようなことが実現できます。
4-1. ⾃然⾔語での論⽂検索
ユーザーは、複雑なPubMed検索構⽂を覚える必要がなく、⾃然⾔語で検索クエリを⼊⼒するだけで、適切な検索が実⾏されます。
例:
- 「糖尿病のインスリン療法に関する2020年以降のRCT」
- 「タイトルにCOVID-19を含むレビュー論⽂」
- 「⼭⽥太郎⽒が著者の2023年の論⽂」
4-2. ⾃動的な翻訳と要約
取得した論⽂のタイトルを⾃動的に⽇本語に翻訳し、アブストラクトを要約します。
これにより、英語が苦⼿な研究者でも、論⽂の内容を素早く把握できます。
4-3. 検索意図に基づく優先度判定
ユーザーの検索意図を考慮して、各論⽂の重要度を⾃動的に判定します。
これにより、⼤量の論⽂の中から、特に重要な論⽂を優先的に確認できます。
4-4. スプレッドシートへの⾃動保存
処理結果をGoogleスプレッドシートに⾃動保存することで、以下のメリットがあります。
- 共有が容易: チームメンバーと簡単に共有できる
- 分析が容易: スプレッドシートの機能を使って、データの分析や可視化が可能
- 履歴管理: 過去の検索結果を蓄積し、後から参照できる
5. シリーズ構成
本シリーズは、以下の5つの記事で構成されています。
| パート | 主な内容 |
|---|---|
| Part 0(本記事): 全体像とPubMed API基礎 | ・ワークフローの全体像 ・PubMed APIの基礎知識 ・このワークフローで実現すること |
| Part 1: パラメータ抽出とE-Search編 | ・ユーザー⼊⼒ノード ・Current Time / パラメータ抽出ノード ・E-SearchとPMID整形 |
| Part 2: E-Fetch / E-Summaryとデータパース編 | ・E-Fetchによる論⽂情報取得 ・変数集約器とXML/JSONパース |
| Part 3: AI処理‧データ整形編 | ・イテレーション処理(並列) ・LLMによる翻訳‧要約‧優先度判定 ・CSV⽣成処理 |
| Part 4: データ保存とGAS連携編 | ・CSV統合とGASへのPOST送信 ・GASコードの詳細解説 |
6. まとめ
本記事(Part 0)では、Difyを使⽤した論⽂検索‧翻訳‧要約ワークフローの全体像と、PubMed APIの基礎知識を解説しました。
次のステップ
次回のPart 1では、⾃然⾔語クエリをPubMed検索パラメータへ落とし込み、E-SearchでPMIDリストを取得するところまでを詳しく解説します。具体的には、以下のノードを実装していきます。
- 開始ノード
- Current Time取得(⽇付確認⽤)
- ⽂章からパラメーター取得(パラメータ抽出ノード)
API⽤リクエストデータ整形(Codeノード) E-Search(HTTP Requestノード)
- PMID配列→⽂字列変換(Codeノード)
これらのノードを実装することで、⾃然⾔語での論⽂検索から詳細データの取得までが⾃動化されます。
シリーズ記事
- Part0(本記事): 全体像とPubMed API基礎
- Part 1: パラメータ抽出とE-Search編
- Part 2: E-Fetchとデータパース編
- Part 3: AI処理‧データ整形編
- Part4: データ保存とGAS連携編

ヘルツレーベンでは、ライフサイエンス業界に特化したDX・自動化支援を提供しています。
PubMedや学術情報の自動収集をはじめ、Slack・Gmailなどを活用したナレッジ共有の仕組みまで、実務に直結するワークフローを設計・導入いたします。
提供サービスの例
- 製薬・医療機器業界での提案活動や調査業務の自動化支援
- アカデミアや研究者向けの文献レビュー・情報共有フローの最適化
- 医療従事者のキャリア開発を支援するリスキリングプログラム
👉 ご興味をお持ちの方はぜひお気軽にお問い合わせください。
お問い合わせフォームはこちら

監修者 株式会社ヘルツレーベン代表 木下 渉
株式会社ヘルツレーベン 代表取締役/医療・製薬・医療機器領域に特化したDXコンサルタント/
横浜市立大学大学院 ヘルスデータサイエンス研究科 修了
製薬・医療機器企業向けのデータ利活用支援、提案代行、営業戦略支援を中心に、医療従事者向けのデジタルスキル教育にも取り組む。AI・データ活用の専門家として、企業研修、プロジェクトPMO、生成AI導入支援など幅広く活動中





