PubMed 最新論文を要約して定期配信 Part2 データベース化
| 目次 |
1. 導入
1.1 本記事の概要
前回(「PubMed最新論文を定期配信 n8nで実現する自動化ワークフロー」)では、PubMed から最新論文を自動取得し、SlackやGmailに通知する仕組みを紹介しました。研究現場において「新着情報をタイムリーに得られる」ことは大きな価値がありますが、それだけでは実務での再利用や体系的な知識管理に繋がりません。
今回の記事では、通知の仕組みをさらに一歩進め、取得した論文をGoogle Sheetsに保存し、重複を排除して整理する方法を解説します。単なる通知から「ナレッジの資産化」へと進化させることで、研究・開発活動における情報活用の基盤が整います。
特に、検索条件を調整することで最新論文だけでなく過去分の文献もまとめて整理でき、結果として疾患領域ごとのナレッジベースが形成されていきます。本記事は、その第一歩を示す実例です。
1.2 想定読者と利用シーン
本記事は以下のような方を対象としています:
- 製薬企業・医療機器メーカーの研究開発・メディカルアフェアーズ部門の担当者
- PubMed検索を日常的に行い、情報を効率的に整理・共有したい方
- Slack通知を既に導入しているが、過去分も含めた体系的なDB化を進めたい方
- 将来的にクラウドDBやBIツールとの連携を視野に入れている方
利用シーンの例
- 「糖尿病」「oncology」といった検索条件で最新+過去数年分を収集し、疾患領域別のナレッジベースを構築
- チームメンバーが即座に新規論文を把握しつつ、既存の知見を検索可能な形で一元管理
- 将来的にBigQueryやSnowflakeへ移行し、自然言語での検索や可視化分析を発展させる
1.3 本記事のポイント
- 通知にとどまらず「ナレッジの資産化」へ進化
- 検索条件を変えることで最新論文だけでなく過去分も体系的に格納可能
- PMIDをキーにした重複排除で効率化
- Slack通知で新規件数を即座に共有
- 将来的にはクラウドDB化により、検索性・分析性・対話型活用を実現
2. 背景と目的
2.1 背景
研究現場では、Slackやメールによる論文通知が一般的に使われています。新着情報を逃さずキャッチできる点は有用ですが、その一方で「流れて終わる」ことがほとんどで、数日後・数週間後に再び参照しようとすると探し出すのが困難になります。
また、Excelやフォルダを使った手動管理も試みられますが、重複や検索性の低下、属人化といった問題が避けられません。これらの手法は、短期的な情報共有には役立っても、長期的な知識基盤の形成には不十分です。
2.2 現状の課題
- 最新論文の把握はできても、過去分を含めた体系的な管理が難しい
- チーム全体での一元管理ができず、必要な文献をすぐに検索できない
- 個人に依存した管理になり、情報の共有や再利用が進まない
2.3 本取り組みの目的
- PubMed論文を自動でDB化し、検索可能な形で格納する
- 検索条件を変えて最新〜過去分まで柔軟に対応
- PMIDを主キーに重複を排除し、効率的に一元管理を実現
- Slack通知で更新件数を即時共有し、チーム内の情報格差を解消
3. n8nの概要
3.1 n8nとは
n8nは、オープンソースのワークフロー自動化ツールです。コードを書かずにさまざまなサービスをつなぐことができ、研究業務でも幅広く活用可能です。
3.2 特徴
- 無料で利用できるセルフホスト型ツール
- Zapierなど商用SaaSと比べて柔軟性が高く、細かな制御や独自ロジックを組み込みやすい
- 医療・研究領域でも、論文モニタリング、DB格納、データ整形など多様な用途に適用可能
実務上の利点
研究現場で使われる他のSaaS(例:Zapier)は便利な反面、費用やデータ管理上の制約があります。n8nはセルフホスト型であるため、医療情報のような機微なデータを扱う場合でも社内環境に閉じて運用できるというメリットがあります。
4. PubMedの概要
4.1 PubMedとは
PubMedは米国国立医学図書館(NLM)が提供する生物医学文献データベースで、世界中の研究者が利用する最重要リソースのひとつです。
4.2 特徴
- 世界最大級の生物医学・ライフサイエンス文献データベース
- 無料で利用可能で、毎日更新される最新情報を取得できる
- APIが公開されており、自動化による情報取得が容易

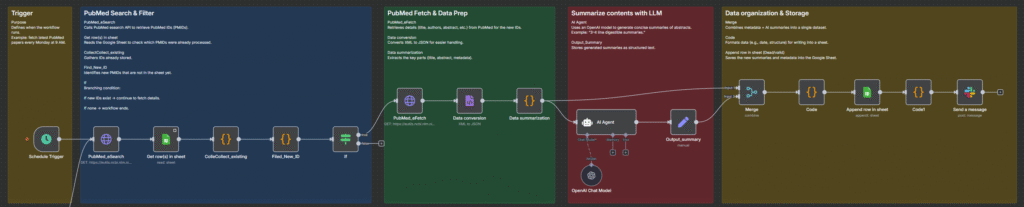
5. PubMed自動化フロー n8nの全体像
今回構築するワークフローは以下の流れで構成されます。
PubMed API → データ整形 → 重複チェック → Google Sheets保存 → Slack通知
- PubMed APIで論文IDを取得
- 既存データと照合し、新規のみ抽出
- 詳細情報を取得して整形
- Google Sheetsに保存
- Slackに件数通知
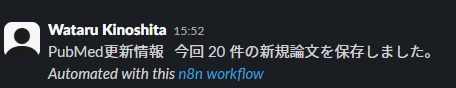
6. 出力イメージ
Slack通知例
Google Sheets保存例
実際の出力は次のようになります。
Google Sheets出力カラムの意味
• Run_date: ワークフローを実行した日付。どのタイミングでDBに登録されたかを記録します。 • Search_query: PubMed検索時に指定した条件(例:「糖尿病」「oncology」など)。後から検索条件ごとに絞り込み・分析するのに役立ちます。 • PMID: PubMedで付与される一意の論文ID。主キーとして扱い、重複排除の基盤になります。 • DOI: 論文のDOI(Digital Object Identifier)。ジャーナル公式サイトなど外部リンクの特定に使用します。 • Title: 論文タイトル。検索や目視確認の基本項目です。 • Authors: 著者名一覧。責任著者・研究グループの把握に利用できます。 • Journal: 掲載ジャーナル名。論文の信頼性や領域特性を把握するための基礎情報です。 • Pub_date: 論文の出版日(または年)。研究の新規性を判断する際に重要です。 • Abstract: 論文抄録。PubMedから取得した原文をそのまま保存しています。 • AI_summary(日本語要約): LLMで生成した要約。短時間で論文の概要を理解でき、非専門者との共有にも有効です。 • URL: PubMed上の論文ページへの直リンク。クリック一つで原文にアクセスできます。
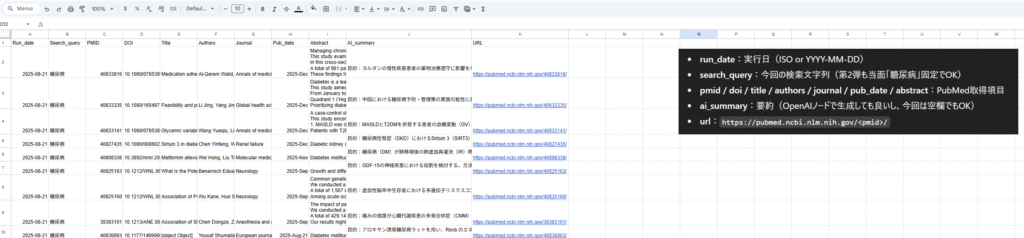
このように列を設計しておくことで、検索条件の記録・AI要約の付与・URLでの直リンクが可能となり、DBとしての価値が大きく高まります。
7. まとめ
本記事では、PubMed論文をGoogle Sheetsに保存し、重複排除とSlack通知を組み合わせるワークフローを紹介しました。
- 通知にとどまらず、ナレッジの資産化が可能になる
- 最新論文だけでなく過去分も含めて整理できる
- 将来的にクラウドDBやBIツールへの移行もスムーズ
研究現場において、単なる情報配信から「知識を蓄積し、活用できる状態に進化させること」は極めて重要です。本記事がその第一歩を踏み出す参考になれば幸いです。
Appendix. 実装ステップ
0) 事前準備(Google Sheets & 列設計)
目的:DB化するための固定スキーマを準備(PMIDは主キー運用)
- Sheet Name: Sheet1(推奨 / gid=0)
- 列(順序固定)
Run_date / Search_query / PMID / DOI / Title / Authors / Journal / Pub_date / Abstract / AI_summary / URL
- n8nで使う識別子
- Document ID:スプレッドシートURLの /d/ と /edit の間
- Sheet Name:Sheet1
- gid:通常 0
1) Trigger
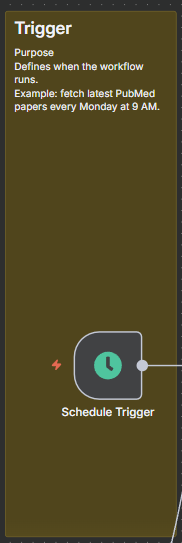
ノード: Schedule Trigger
Purpose: 本番の定期実行(例:毎週月曜 10:00 JST)
mode: everyWeek dayOfWeek: Monday hour: 10 # Tokyo
ノード: Manual Trigger(When clicking ‘Execute workflow’)
Purpose: 開発・スポット実行用
接続: → PubMed_eSearch
2) PubMed Search & Filter
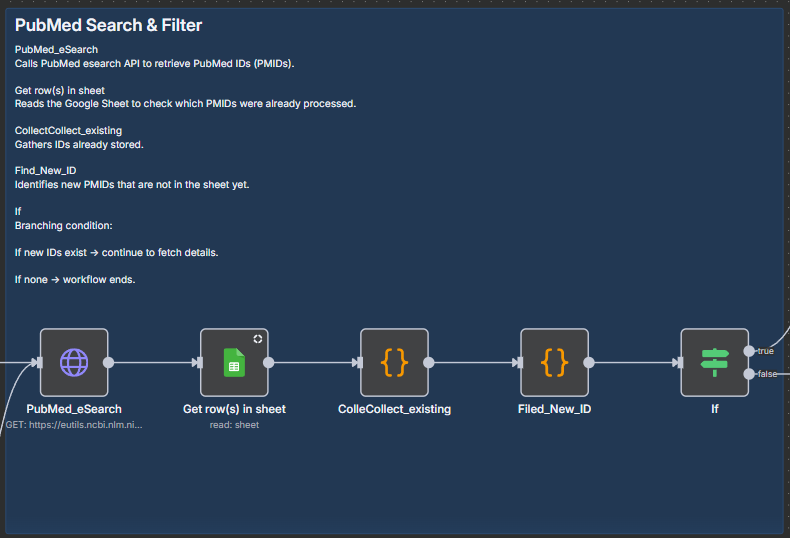
ノード: PubMed_eSearch(HTTP Request)
Purpose: PubMed eSearch APIでPMID一覧を取得
Method: GET URL: https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi
Query Parameters:
db=pubmed term=diabetes[Title/Abstract] OR "diabetes mellitus"[MeSH Terms] datetype=pdat reldate=7 retmax=20 sort=pub+date retmode=json
過去分バッチ取得の例
- reldate を使わず期間指定:
datetype=pdat&mindate=2022/01/01&maxdate=2024/12/31
- もしくは term に期間を埋め込み:
term=(diabetes[Title/Abstract]) AND ("2022"[Date - Publication] : "2024"[Date - Publication])
ノード: Get row(s) in sheet(Google Sheets: Read)
Purpose: 既存PMID(C列)を読み出す
• Document ID:<あなたのID> • Sheet:Sheet1(gid=0) • Range例:C2:C10000
ノード: ColleCollect_existing(Code)
Purpose: C列から既存PMID配列 seen を作成
// 入力: Google Sheets Read の出力
const rows = $items();
const seen = rows
.map(r => String(r.json.PMID ?? r.json.C ?? '').trim())
.filter(x => x);
return [{ json: { seen } }];
ノード: Filed_New_ID(Code)
Purpose: eSearchの idlist から未登録PMIDのみ抽出
// 入力: PubMed_eSearch の idlist と ColleCollect_existing の seen を前段で合流させておく
const ids = $input.first().json.idlist || []; // eSearchのPMID配列
const seen = $input.item(1)?.json.seen || []; // 2番目入力: 既存PMID
const newIds = ids.filter(id => !seen.includes(String(id)));
return [{ json: { ids: newIds } }];
ノード: IF
Purpose: 新規がある時のみ詳細取得へ
条件:
$json.ids && $json.ids.length > 0
- True → 次のセクションへ
- False → ワークフロー終了(新規なし)
3) PubMed Fetch & Data Prep
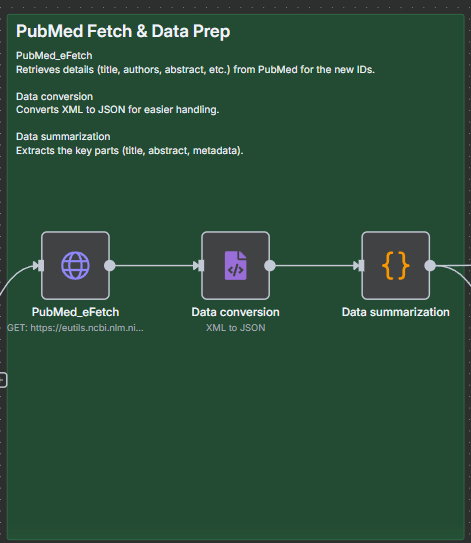
ノード: PubMed_eFetch(HTTP Request)
Purpose: 新規PMIDの詳細XMLを取得(タイトル/著者/抄録/誌名/発行日/DOI)
Method: GET URL: https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi
Query Parameters:
db=pubmed
id={{ $json.ids.join(',') }}
retmode=xml
ノード: Data conversion(XML)
Purpose: XML → JSON 変換(オプションは既定でOK)
ノード: Data summarization(Code)
Purpose: JSONの揺れを吸収しつつ主要フィールドを正規化して1論文=1アイテムに
// 入力: XMLノードの出力(JSON化済)
const set = $json.PubmedArticleSet || {};
const list = set.PubmedArticle || [];
function txt(v){ return (v?._text ?? v?.['#text'] ?? v ?? '').toString(); }
function joinArray(a){ return Array.isArray(a) ? a.map(x => txt(x)).join(' ') : txt(a); }
return list.map(p => {
const mc = p.MedlineCitation || {};
const art = mc.Article || {};
const pmid = txt(mc.PMID);
// DOI(ELocationIDに入ることが多い/複数のとき先頭を採用)
let doi = '';
const el = art.ELocationID;
if (Array.isArray(el)) {
const cand = el.find(x => txt(x).includes('10.'));
doi = cand ? txt(cand) : '';
} else if (txt(el).includes('10.')) {
doi = txt(el);
}
const title = joinArray(art.ArticleTitle);
const journal = txt(art.Journal?.Title);
// 年 or 年月日を安全に抽出(Year, MedlineDateなど揺れに対応)
const pub = art.Journal?.JournalIssue?.PubDate || {};
const pubYear = txt(pub.Year) || (txt(pub.MedlineDate).match(/\d{4}/)?.[0] ?? '');
const abstract = joinArray(art.Abstract?.AbstractText);
const authors = (art.AuthorList?.Author || [])
.map(a => `${txt(a.LastName)} ${txt(a.ForeName)}`.trim())
.filter(x => x && x !== '')
.join(', ');
const url = pmid ? `https://pubmed.ncbi.nlm.nih.gov/${pmid}/` : '';
return {
pmid,
doi,
title,
authors,
journal,
pub_date: pubYear,
abstract,
url,
};
});
4) Summarize contents with LLM
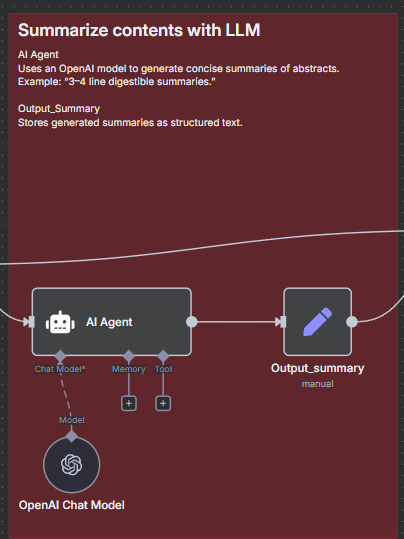
ノード: OpenAI Chat Model(もしくは AI Agent → 内部でChat Model)
Purpose: 抄録を150–220字の日本語で要約、タグ付与
System Message
あなたは医学論文の抄読支援者です。 出力は必ず日本語。誇張せず事実ベースで要約し、曖昧な点は「不明」と書く。 記載のない情報を推測しない。
User Prompt
次のPubMed論文を150~220字で日本語要約してください。
必ず「目的 / 方法 / 主な結果 / 含意」を短く含め、数値はあれば保持。
最後に【タグ】を1~2語で付与。
タイトル: {{$json.title}}
抄録: {{ $json.abstract }}
雑誌: {{$json.journal}}
発行日: {{$json.pub_date}}
URL: {{$json.url}}
ノード: Output_summary(Code)
Purpose: LLM出力のキー差を吸収して summary_ja に正規化
const c = $json?.choices?.[0]?.message?.content ?? $json?.content ?? $json?.text ?? '';
return [{ json: { summary_ja: c } }];
5) Data organization & Storage
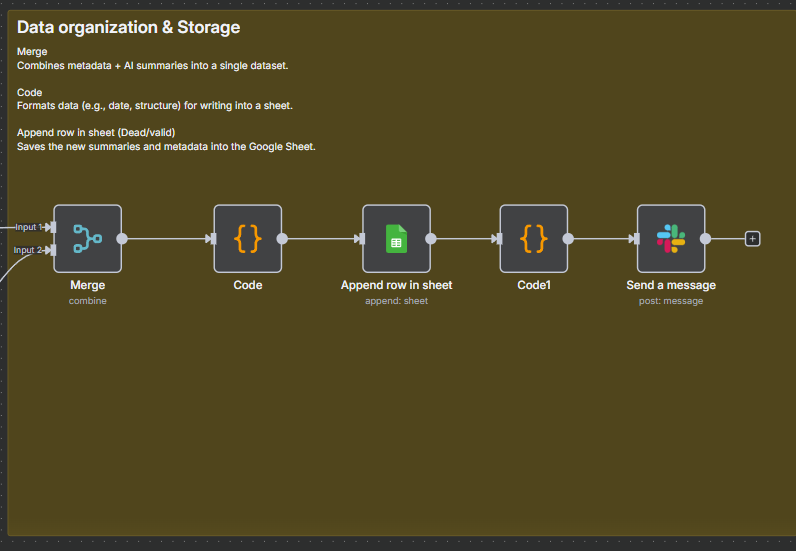
ノード: Merge(Mode: combine / combineBy: combineByPosition)
Purpose: メタデータ(Data summarization)と要約(Output_summary)を1:1で結合
ノード: Code(整形)
Purpose: 最終マッピング用にキー名を統一、監査メタを付与
const now = new Date();
const pad = n => String(n).padStart(2,'0');
const run_date = `${now.getFullYear()}-${pad(now.getMonth()+1)}-${pad(now.getDate())}`;
return [{
json: {
Run_date: run_date,
Search_query: $json.search_query ?? '糖尿病', // 必要に応じ固定/可変
PMID: $json.pmid,
DOI: $json.doi,
Title: $json.title,
Authors: $json.authors,
Journal: $json.journal,
Pub_date: $json.pub_date,
Abstract: $json.abstract,
AI_summary: $json.summary_ja,
URL: $json.url
}
}];
ノード: Append row in sheet(Google Sheets: Append)
Purpose: シートに追記(列名→値のマッピングは上記キーと一致)
- Document ID:<あなたのID>
- Sheet:Sheet1(gid=0)
- 列マッピング例:
o Run_date → {{$json.Run_date}}
o Search_query → {{$json.Search_query}}
o PMID → {{$json.PMID}}
o DOI → {{$json.DOI}}
o Title → {{$json.Title}}
o Authors → {{$json.Authors}}
o Journal → {{$json.Journal}}
o Pub_date → {{$json.Pub_date}}
o Abstract → {{$json.Abstract}}
o AI_summary → {{$json.AI_summary}}
o URL → {{$json.URL}}
運用堅牢化:PMID列にデータ検証(重複禁止)または UNIQUE 関数を設定しておくと二重実行時の重複も抑止できます。
ノード: Code1(Code)
Purpose: 今回追加できた件数を集計
return [{ json: { new_count: items.length } }];
ノード: Send a message(Slack: post message)
Purpose: チャネルに結果通知
PubMed更新情報
今回 {{ $json["new_count"] }} 件の新規論文を保存しました。
条件を変えながら格納するレシピ(運用Tips)
• 領域変更:term=oncology[Title/Abstract] AND Japan[Affiliation] • 期間指定:datetype=pdat&mindate=2020/01/01&maxdate=2025/08/29 • 大量取得:retmax=200 と retstart=0,200,400...、または usehistory=y を併用

ヘルツレーベンでは、ライフサイエンス業界に特化したDX・自動化支援を提供しています。
PubMedや学術情報の自動収集をはじめ、Slack・Gmailなどを活用したナレッジ共有の仕組みまで、実務に直結するワークフローを設計・導入いたします。
提供サービスの例
- 製薬・医療機器業界での提案活動や調査業務の自動化支援
- アカデミアや研究者向けの文献レビュー・情報共有フローの最適化
- 医療従事者のキャリア開発を支援するリスキリングプログラム
👉 ご興味をお持ちの方はぜひお気軽にお問い合わせください。
お問い合わせフォームはこちら





